
引言
人类基因组计划大规模测序工作的完成标志生命科学的研究将进入后基因组时代。由于蛋白质是生理功能的执行者以及生命现象的体现者,对蛋白质功能的研究将成为后基因时代研究的核心内容之一.
早期的蛋白质功能预测方法大多基于蛋白质序列间的同源性,认为序列上相似的蛋白质具有相似的功能。通过运用FASTA、BLAST等工具对数据库中已知功能的蛋白质序列进行搜索,为待注释功能的蛋白质寻找序列相似性最高的蛋白质来进行功能预测.随着高通量实验技术的应用,基于蛋白质交互网络的蛋白质功能预测方法受到越来越多的关注。
Deng 假设蛋白质的功能独立于与其直接相邻的邻居之外的所有蛋白质,提出了基于马尔可夫随机场模型来对蛋白质进行功能注释,并且取得了不错的预测效果。本文在Deng模型基础之上将其推广到二阶马尔可夫随机场模型,并且利用伪似然估计方法来估计模型参数,在平均预测准确率的意义下,取得了比Deng模型更好的预测结果。
1.二阶马尔可夫随机场模型
我们如下定义蛋白质的二阶邻居:对于一个蛋白质,它的所有直接交互伙伴以及它所有直接交互伙伴的直接交互伙伴都称为该蛋白质的二阶邻居(不包括自身)。为了简化计算,我们假设二阶邻居中的所有蛋白质对预测蛋白质功能的影响是相同的。
假 设 一 个 基 因 组 有 N 种 蛋 白 质 P1,?,PN以 及 M 类 功 能F1,?FM.对于每一个蛋白质 Pi,我们定义Nei(i)为它的邻居组成的蛋白质集合。记πj表示所有的蛋白质有 Fj功能的比例。假设 S 是所有交互蛋白质对组成的集合。对于某个感兴趣的功能,设(x1,?,xN)是 N个蛋白质的功能标记:如果第 i 个蛋白质有这个功能,则 xi=1;否则xi=0.根据Deng模型我们知道第 i 个蛋白质有此功能的先验概率为:【1】
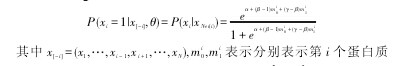
交互邻居中无此功能和有此功能的个数,α=ln?è??π1-π, β-1和 γ-β为待定参数。我们利用计算机视觉中常用的伪似然估计方法来估计,具体操作如下:
首先考虑如下的条件似然函数:【2-3】


选取适当的初值和阈值后,便可以估计出参数 β-1和 γ-β,从而可以得到 S 中每个蛋白质含有任意一种功能的后验概率。
2.实验结果及分析
在本文中,我们用平均预测准确率来衡量模型的优劣。换言之,平均预测准确率越高,模型性能越好,反之则性能越差。这里的平均预测准确率是按如下定义的:
对每种功能来讲,首先根据模型计算出 S 中每个蛋白质含有这种功能的概率,然后取阈值为0.5,如果某蛋白质含有此功能的后验概率大于(或等于)0.5,我们便认为该蛋白质拥有这个功能。于是得到 S 中蛋白质的预测功能矩阵,将其与原始数据相比较,预测正确的个数比上S 中总的蛋白质个数便是此功能的预测准确率,所有功能的预测准确率的算术平均即为平均预测准确率。
本文中的实验是在以下软硬件平台的PC机下完成:CPU:T6570,主频率2.1G,2G内存,Windows7操作系统,编译工具是Matlab.其中酵母菌蛋白质数据估计参数 β -1和 γ-β时初值分别选择为-0.1和1,阈值选择为 0.001.Deng 模型和本文模型的平均预测准确率分别为 96.18%、97.26%,从而在平均预测准确率的意义下,本文模型要优于 Deng 模型。
3.总结
针对蛋白质的功能预测问题,本文基于二阶马尔可夫随机场模型对蛋白质进行了功能预测。结果表明,在平均预测准确率意义下,取得了较好的预测效果。值得提出的是,本文假设只有蛋白质的二阶邻居对其功能有影响且影响是相同的,而实际上不同的二阶邻居对其功能的影响是不尽相同的,并且蛋白质交互网络中距离更远的蛋白质也可能对其功能有影响,这些都是需要后续研究的问题。
参考文献
[1]朱薿。蛋白质相互作用网络在蛋白质功能预测中的应用[J].咸宁学院学报(医学版),2008,22(4):364-366.
[2]Pearson W. R, Lipman D. J. Improved Tools for BiologicalSequence Comparison[J]. Proc Natl Acad Sci USA, 1988, 85(8) :2444-2448.
[3]Altschul S. F, Madden T. L, Schaffer A. A, et al. GappedBLAST and PSI- BLAST: a New Generation of Protein DatabaseSearch Programs[J]. Nucleic Acids Res, 1997, 25(17) : 3389-3402.
[4]Deng M, Zhang K, Mehta S, et al. Prediction of ProteinFunction Using Protein- protein Interaction Data[J]. J Comput Biol,2003, 10(6): 947-960.
[5]Li S. Z Markov Random Field Modeling in Computer Vi-sion[M]. Springer-Verlag: Tokyo. 1995.





