摘 要: 近年来出现的几种以人工核酸酶为代表的新型基因编辑技术, 包括锌指蛋白核酸酶 (zinc-finger nuclease, ZFN) 技术、转录激活因子样效应核酸酶 (transcription activator-like effector nuclease, TALEN) 技术、成簇的规律间隔的短回文重复序列/CRISPR相关蛋白 (clustered regularly interspaced short palindromic repeats/CRISPR associated proteins, CRISPR/Cas) 系统, 在科学研究以及家畜育种等方面已被广泛应用。这些基因编辑技术是通过特异性结构识别靶位点, 核酸酶发挥切割作用, 对靶位点进行定点编辑, 具有高效准确、制作简单等特点。本文阐述了上述3种新型基因编辑技术的基本原理, 比较了不同基因编辑技术的差异, 总结了在主要家畜猪、牛、羊上的应用, 提出目前这三种基因编辑技术存在的主要问题, 并对这些技术的应用前景进行了展望。
关键词: 基因编辑; ZFN; TALEN; CRISPR/Cas; 家畜;
Abstract: Several novel gene editing techniques have been applied in scientific research and livestock breeding in recent years, including zinc finger protein nuclease (ZFN) , transcriptional activator-like effector nuclease (TALEN) , clustered regularly interspaced short palindromic repeats/CRISPR associated proteins (CRISPR/Cas) system. These gene editing techniques site-directly edit genomes by recognizing the target sites according to the specific structure followed by endonuclease dissection, which is characterized by high efficiency, accuracy, and simple production. In this paper, we demonstrated the basic principles, differences and potential main problems of above three gene editing techniques, summarized the application in the main livestock pigs, cattle and sheep, and provided an outlook of the future of these techniques in application.
Keyword: Gene editing; ZFN; TALEN; CRISPR/Cas; Livestock;
基因编辑技术是指人为地对目的基因进行移除和定点插入的操作, 从而实现特定基因的敲除和敲入等。基因编辑技术可以实现在细胞水平, 器官水平, 以及个体水平进行精确而有效的基因组编辑, 从而被广泛应用于生物技术领域。
在过去的数十年中, 一系列通过核酸内切酶介导的新型基因编辑技术被开发出来, 主要包括锌指核酸酶 (zinc-finger nuclease, ZFN) 技术, 转录激活因子样效应核酸酶 (transcription activator-like effector nuclease, TALEN) 技术和成簇的规律间隔的短回文重复序列相关核酸酶 (clustered regularly interspaced short palindromic repeats/CRISPR associated proteins, CRISPR/Cas) 系统。这三种核酸酶基于由序列特异性的DNA结合结构域与非特异性的DNA切割结构域组成 (Carroll, 2011;Urnov et al., 2010) 。他们通过特异性结合DNA序列, 产生双链断裂 (double-strand breaks, DSBs) , 启动细胞自身DNA修复机制, 以非同源末端连接 (non-homologous end joining, NHEJ) 或者同源重组修复 (homology-directed repair, HDR) 的方式进行基因高效精确编辑 (Wyman, Kanaar, 2006) 。这三种新型基因编辑技术已经在细菌 (Shipman et al., 2017) 、酵母 (Saccharomyces cerevisiae) (Xie et al., 2018) 、人类 (Homo sapiens) 细胞 (Verma et al., 2017) 、食蟹猴 (Macaca fascicularis) (Yao et al., 2018) 、果蝇 (Drosophila melanogaster) (Denecke et al., 2017) 、斑马鱼 (Barchydanio rerio) (Zhang et al., 2017) 、爪蟾 (Xenopus laevis) (Delay et al., 2017) 、小鼠 (Mus musculus) (Nakasuji et al., 2017;Roper et al., 2018) 、拟南芥 (Arabidopsis thaliana) (Ryder et al., 2017) 、水稻 (Oryza sativa) (Yin et al., 2017) 、小麦 (Triticum aestivum) (Shan et al., 2013) 等多种生物体中得到了广泛的研究和应用。在三种主要家畜动物猪 (Zheng et al., 2017) 、牛 (Gao et al., 2017;Wu et al., 2015) 、羊 (葛恒涛, 2016;朱红梅, 2016) 上的应用, 近些年也取得了很大的进展。
1 新型基因编辑技术介绍
1.1 ZFN技术
ZFN又称锌指蛋白核酸酶 (zinc-finger protein nuclease, ZFPN) , 是一种经过人工改造的核酸内切酶。科学工作者1983年首次在非洲爪蟾卵母细胞的转录因子ⅢA中发现了与DNA具有特异性结合亲和力的锌指蛋白 (zinc finger protein, ZFP) (Mani et al., 2005) 。其在20世纪90年代末首次得到研究和应用 (Bibikova et al., 2003) , 从而实现了基因的高效定点编辑, 开创了基因修饰的先河。

ZFN其结构主要由两部分组成, 即ZFP构成的特异性DNA结合域 (DNA-binding domain) 和DNA切割域 (DNA cleavage domain) (Kim et al., 1996) 。DNA切割域由限制性内切酶FokⅠ演变而来, DNA结合域用于识别特定的DNA序列, 位于ZFN的N末端。含有大约30个氨基酸残基的锌指 (Zinc-finger, ZF) 是构成ZFP结构域的DNA结合基序 (DNA-binding motif) (Klug, 2010) 。可以将多个锌指结构组装到一起, 用以识别更长的DNA序列。因此, 通常将3~6个Cys2His2类型的锌指结构相结合, 组装构成ZFP结构域, 其在DNA链上沿着3'到5'的方向与靶序列相结合 (Gaj et al., 2012) 。FokⅠ切割域位于ZFN的C末端, FokⅠ核酸酶发现于海床黄杆菌 (Flavobacterium okeanokoites) , 其在细菌体内通过二聚化发挥核酸内切酶的活性, 使目的基因位点产生双链断裂 (Palpant, Dudzinski, 2013) 。一对ZFN的两个单体分别由其3'到5'以及5'到3'方向识别靶标DNA双链, 两个FokⅠ核酸酶切割域发挥切割作用, 特异性的打靶目的基因 (Hauschild-Quintern et al., 2013) 。锌指核酸酶使用的关键是构建识别不同碱基的ZFP, 通过优化氨基酸的组成来达到基因精确编辑的目的。
在基因编辑过程中, 把锌指核酸酶的质粒载体导入细胞后, 载体上的核定位信号将会引导锌指核酸酶进入细胞核, 锌指DNA结合域将与目标序列发生特异性结合, FokⅠ切割域于两个结合位点的间隔区切割产生DNA双链断裂 (Carroll, 2011) , 细胞可通过HDR或NHEJ方式产生基因敲除、点突变、基因敲入, 从而实现基因的定向修饰操作 (Kim, Kim, 2011) 。
1.2 TALEN技术
TALEN也是一种人工合成的核酸酶, 1989年转录激活样效应因子 (trancription activator-like effector, TALE) 首次在感染植物的一种细菌—黄单胞菌 (Xanthomonas) 中被发现 (Bonas et al., 1989) 。TALE通过细菌Ⅲ类分泌系统进入细胞, 通过与特异基因启动子结合来调节转录, 促进细菌的集落形成。科学工作者将TALE具有的特异性结合能力与FokⅠ核酸酶具有的切割能力相结合, 制作出了具有特异性基因组编辑能力的工具TALEN, TAL-EN技术由此产生, 并被迅速用于生物技术方面的研究, 逐渐取代了ZFN技术 (Bedell et al., 2012) 。
与ZFN相类似, TALEN也由两部分组成。一部分是TALE蛋白DNA结合结构域, 另一部分是与ZFN相同的FokⅠ核酸酶切割域, 前者负责目标序列的特异性识别, 后者进行靶位点的切割 (Moscou, Bogdanove, 2009) 。TALE由N端转运信号、C端核定位信号、转录激活结构域和DNA结合结构域构成 (Deng et al., 2012;Ul et al., 2015) 。DNA结合结构域由多个重复序列组成, 每个重复序列由33~35个氨基酸组成, 其模块内的第12和13位的氨基酸具有两个多态性, 称为双氨基酸残基 (repeat variable diresidues, RVD) (Osborn et al., 2013) 。鉴于该DNA结合结构域的模块化特性, 可以将具有不同特异性的RVD组装成阵列以靶向特异性的DNA序列。FokⅠ核酸酶与TALE的C端相连, 通过识别特异的DNA序列, 对其进行定点切割, 引起DSBs, 并且启动细胞自我修复机制, 以HDR或NHEJ的方式, 实现对特定基因的靶向修饰, 包括基因敲除、敲入和基因定点突变等 (Hinkley et al., 2011;Li et al., 2011) 。Carlson等 (2012) 将TALEN技术应用于猪 (Sus scrofa) 和牛 (Bos taurus) 的胚胎基因组编辑, 引起家畜基因组的的特定遗传修饰, 包括单等位基因敲除、双等位基因敲除以及基因敲入等等, 改变了家畜物种的遗传学组成和基因组的结构和功能。TALEN基因编辑技术是一种崭新的分子生物学工具, 被认为是基因编辑技术发展史上的里程碑, 是继ZFN之后的第二代基因编辑技术, 被《Science》杂志评选为2012年度十大科学突破的新技术。
1.3 CRISPR/Cas9技术
Ishino等 (1987) 在大肠杆菌 (Escherichia coli) 中发现了成簇的规律间隔的短回文重复序列, 到2012年, 科学家才正式命名为CRISPR。后来又一种经过人工改造的核酸内切酶, 即CRISPR/Cas技术, 迅速在动物、植物、微生物上得到广泛应用 (Nakasuji et al., 2017;Ryder et al., 2017;Xie et al., 2018) 。
CRISPR/Cas系统两部分构成, 即CRISPR和Cas核酸酶。CRISPR是指成簇的规律间隔的短回文重复序列 (Mojica et al., 2000) , 其序列由一个前导区 (leader) 、多个短的重复序列区 (repeat) 以及多个间隔区 (spacer) 构成 (Makarova et al., 2006) 。leader长度通常在400 bp左右, 其序列上富含A、T碱基, 负责启动CRISPR序列的转录 (Sorek et al., 2008) 。repeat形成发卡结构, 含有长度为22~48 bp的回文序列 (Grissa et al., 2007) 。长度为26~73 bp的spacer将各个重复序列隔开 (Deltcheva et al., 2011;Karginov, Hannon, 2010) 。Cas核酸酶即CRISPR相关蛋白, 现在基因工程中最常用的核酸酶是Cas9蛋白, 该蛋白是一种多结构域核酸酶蛋白 (Mojica, Montoliu, 2016) 。
CRISPR/Cas系统主要被分为三类:Ⅰ型、Ⅱ型和Ⅲ型。目前, 被广泛应用的是来源于Ⅱ型的CRISPR/Cas9系统由Cas9核酸内切酶、CRISPR RNA (crRNA) 、反式激活crRNA (tracrRNA) 组成。Cas9核酸酶与两种RNA成分形成复合物, crRNA在目标识别中发挥作用, 而tracrRNA在复合物形成过程中在crRNA成熟和Cas9蛋白的稳定中发挥重要的作用 (Jinek et al., 2012) 。crRNA和tracrRNA均具有非编码能力并且彼此部分互补 (Feng et al., 2013) , 两种RNA组分形成一种称为单向导RNA (sgRNA) 的嵌合RNA (Doudna, Charpentier, 2014) 。Cas9核酸酶具有两个独立的Ruvc和HNH结构域, 当CRISPR/Cas9复合物与PAM (protospacer adjacent motif) 序列结合时, 这些结构域都被激活, HNH参与互补链的切割, 而Ruvc参与切割非互补链, 两条链在重新接合后导致移码突变或插入缺失 (Jinek et al., 2012) 。Csa9核酸酶行使功能最重要的区域是PAM序列区域, PAM序列涉及CRSIPR/Cas9复合物与靶区域的早期关联, PAM基序的序列通常是NGG, 且存在于靶序列的下游区域 (Ran et al., 2015) 。现在, CRISPR系统组件已经简化为两部分, 即Cas9核酸内切酶和sgRNA (Doudna, Charpentier, 2014;Jinek et al., 2012;Ma et al., 2016) 。
CRISPR/Cas系统的发展日新月异, 呈现了多元化发展的趋势。2015年美国麻省理工学院张锋团队发现了类似于Cas9但结构更简单的核酸酶—Cpf1, Cpf1不需要核糖核酸酶 (RNaseⅢ) 和tracrRNA便可独自完成对crRNA的加工成熟过程 (Zetsche et al., 2015) 。该研究团队还发现了一种只靶向和切割细菌内特异RNA序列的新型蛋白—C2c2 (Abudayyeh et al., 2016) 。C2c2作为一种新发现的针对RNA的分子生物学工具, 将被用于RNA的功能、运动和定位研究中。此外, 美国哈佛大学David Liu实验室2016年首次发现了基于胞嘧啶脱氨酶与CRISPR/Cas9融合形成的单碱基编辑技术 (base editor, BE) , 该技术实现了胞嘧啶 (cytosine, C) 到胸腺嘧啶 (thymine, T) 的单碱基转换 (Komor et al., 2016) 。2017年, 该实验室再次取得重要进展, 建立了新的单碱基编辑系统, 实现了腺嘌呤 (adenine, A) 到鸟嘌呤 (guanine, G) 的精确转换 (Gaudelli et al., 2017) 。这两套系统在不引入DNA双链断裂也不需要重组修复模板的情况下可以实现对基因组更加安全、高效、精准的单碱基编辑, 而且其效率远远高于DSBs引起的HDR修复方式 (Gaudelli et al., 2017;Komor et al., 2016) 。近日, 伊利诺伊大学的一个研究团队提出了一种称为CRISPR-SKIP的通用方法, 该方法利用胞嘧啶碱基编辑器 (cytosine base editor, CBE) 控制基因剪接, 实现了靶基因处的外显子跳跃。这意味着, CRISPR-SKIP可以在不引入DSBs情况下来消除突变的基因序列, 还能影响基因的表达和调控方式, 有望治疗多种遗传性疾病 (Gapinske et al., 2018) 。
随着第三代基因编辑技术CRISPR/Cas系统的横空出世, 立刻成为了基因编辑历史上的又一重要的里程碑。该技术克服了传统的基因编辑技术的操作周期长、打靶效率低、应用范围窄的缺点。CRISPR/Cas系统的不断研发和多元化发展拓展了该技术的应用领域, 并且使该技术用起来更完美, 更简单, 效率更高, 脱靶效应大大降低, 安全性更好, 成为目前基因编辑技术的主流方法和工具。
2 三种新型基因编辑技术的优劣
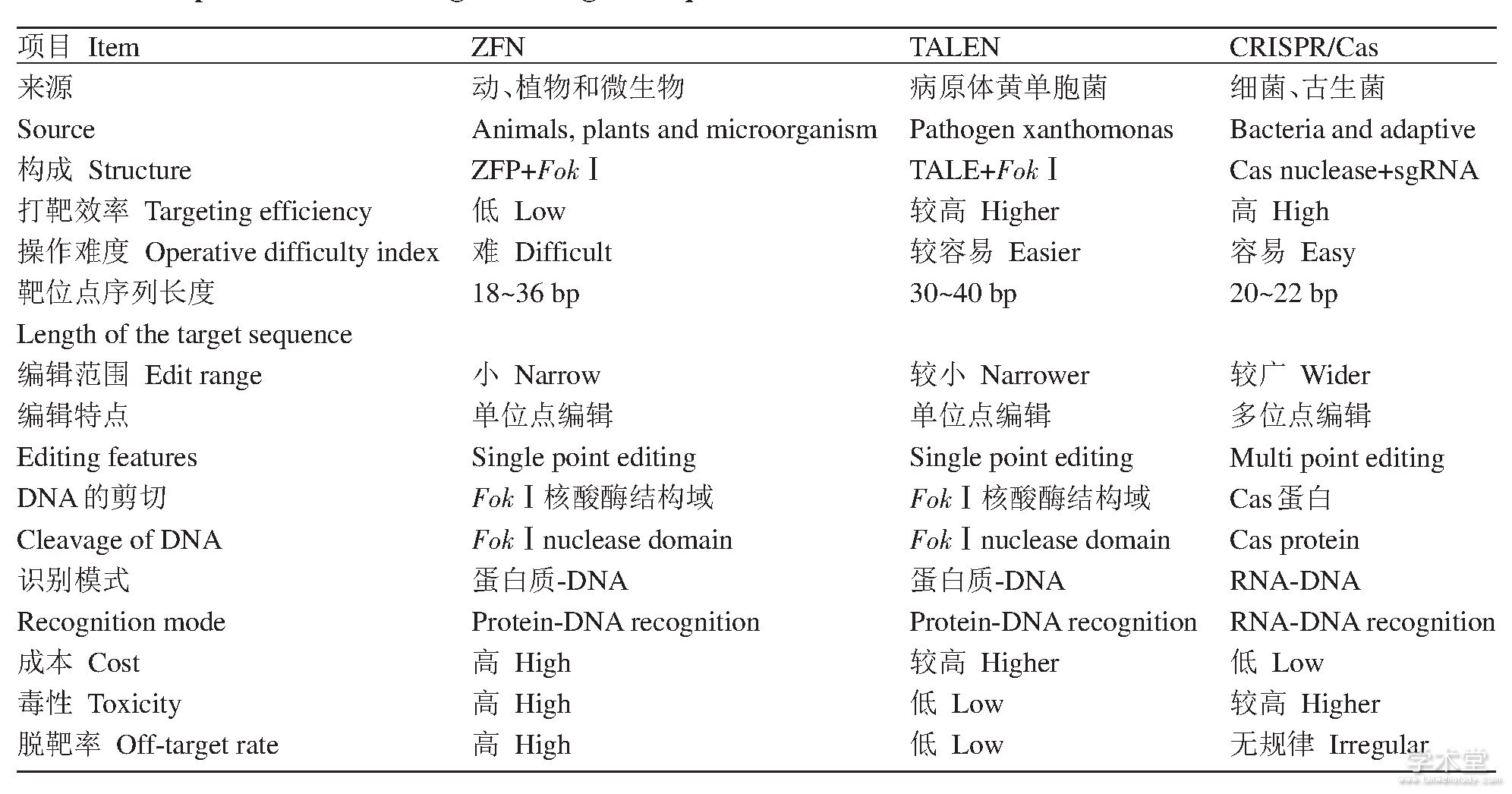
三种新型基因编辑技术 (ZFN, TALEN和CRISPR/Cas) 均能实现基因组的定点修饰和编辑, 应用范围有很大程度的重合, 但是这三种技术在使用方面有不同的技术特点和适用范围 (表1) 。ZFN技术最先发现, 研究应用时间比较长, 技术较为完善, 但ZFN的识别结构域中存在广泛的上下文依赖效应, 在对ZFN的选择方面还有很大的难度, 且成本较高, 产生较强的细胞毒性, 容易引起基因组中其他基因的突变以及染色体畸变 (Pattanayak et al., 2011;Radecke et al., 2010) 。TALEN技术相比ZFN技术, 其识别位点及选择范围变得较宽, 而且成本大大降低, 打靶效率显着提高和细胞毒性显着降低, 但是其限制因素是在识别位点前的第一个碱基必须是胸腺嘧啶, 且构建TALEN模块比较复杂, 费时费力。最近几年兴起的CRISPR/Cas技术, 其打靶效率和适用性要比TALEN和ZFN技术高很多, 但是也不可避免的存在一些不足, 目前主要存在的问题是PAM序列的依赖性与脱靶效应 (Fu et al., 2013;Hsu et al., 2013) 。
3 基因编辑技术在家畜上的应用
3.1 在家畜抗病育种中的应用
家畜疾病频发给养殖业带来了巨大的经济损失, 是目前制约畜牧业发展的重要因素;人畜共患病的爆发同样威胁着人类的安全。此外, 家畜疾病还带来了一系列诸如环保、抗生素残留以及食品安全等问题。人们逐渐意识到, 单纯依靠防疫和药物治疗已无法彻底防控家畜疾病。近些年, 科学工作者将新型基因编辑技术应用于家畜抗病育种中去, 取得了很大的进展。
表1 三种新型基因编辑技术的比较Table 1 Comparison in three new gene editing techniques
Richt等 (2007) 运用传统的同源重组技术在牛体内敲除了朊蛋白基因 (prion protein gene, PRNP) , 使其脑部的PrPC蛋白表达显着下调, 可以防止由朊蛋白造成的疯牛病 (Mad cow disease) 的发生。笔者所在实验室利用ZFN技术将外源的人溶菌酶基因和溶葡萄球菌素基因定点插入到牛酪蛋白位点 (CSN2) , 通过克隆技术获得了对乳腺炎具有抗性的奶牛 (Liu et al., 2013;Liu et al., 2014) 。同时, 本实验室还利用TALEN和CRISPR/Cas9技术, 将外源的SP110基因和NRAMP1基因定点插入到牛基因座内, 生产出具有抗结核病功能的转基因牛, 研究结果不仅对牛结核病的防御与控制有重大意义, 同时为动物抗病育种提供新思路 (Gao et al., 2017;Wu et al., 2015) 。有研究发现, 猪CD163蛋白是猪繁殖与呼吸综合征 (porcine reproductive and respiratory syndrome, PRRS) 病毒感染猪肺泡巨噬细胞的主要受体 (Calvert et al., 2007) 。美国密苏里大学Whitworth、英国爱丁堡大学罗斯林研究所Burkard以及浙江大学王少华等分别利用CRISPR/Cas9技术成功获得了CD163基因敲除猪, 对PRRS病毒具有显着抗性, 猪繁殖与呼吸综合征病毒 (Porcine reproductive and respiratory syndrome virus, PRRSV) 抗病猪的产生对于人类对抗对养猪业具有致命性打击的繁殖与呼吸综合征的发生具有重要意义 (Burkard et al., 2017;Whitworth et al., 2015;王少华等, 2018) 。
3.2 在家畜生产性状改良中的应用
肌肉生长抑制素基因 (myostatin, MSTN) 是肌肉生长的负调控因子, 该基因的表达可抑制肌细胞的增殖与分化, 对该基因进行突变有利于肌肉的生长发育, 可以显着增加肌纤维的直径、提高肌纤维数量 (Mcpherron, Lee, 1997) 。Luo等 (2014) 利用锌指核酸酶技术首次生产出MSTN基因敲除的牛, 这些牛表现出显着的双肌表型, 大大提高了肉牛的产肉量, 为高瘦肉率肉牛、肉羊等肉用家畜新品种培育奠定基础。Wang (2015) 年报道应用CRISPR/Cas9技术, 将sgRNA和Cas9RNA共注射到绒山羊 (Capra hircus) 原核期胚胎, 经胚胎移植生产出了同时敲除FGF5基因和MSTN基因的山羊, 山羊被毛长度显着增加, 且呈现双肌表型, 实现了在山羊体内同时敲除多个基因, 加快了优质绒山羊的培育。在Yu等 (2016) 利用TALEN技术结合体细胞核移植技术靶向敲除了山羊体内的MSTN基因, 生产出了三只发育良好的敲除MSTN基因的山羊, 说明了TALEN技术同样可以对山羊的基因组进行定向编辑, 为TALEN技术应用于山羊的育种提供了可能。Wang等 (2016) 利用CRISPR/Cas9系统结合原核注射技术在绵羊 (Ovis aries) 体内同时实现了MSTN、刺鼠信号蛋白基因 (agouti signal protein, ASIP) 和β-胡萝卜素加氧酶2基因 (beta-carotene oxygenase 2, BCO2) 三基因靶向敲除, 且在潜在的脱靶位点未发现脱靶效应, 表明CRISPR/Cas9技术可以同时针对多个基因进行可遗传修饰, 且有较低的脱靶效应, 达到有效改善家畜生产性能的目的。此外, 还有多家单位利用CRISPR/Cas9系统获得了MSTN基因敲除的家畜动物 (Guo et al., 2016;Wang et al., 2017) , 并积极用于扩繁育种建群。
不饱和脂肪酸ω-3有益于调整动物机体内部脂类代谢, ω-3脂肪酸脱氢酶表达由Fat-1基因调控, 使脂肪酸由ω-6形式转变为ω-3。张驹等通过CRISPR/Cas9技术成功敲除了山羊的MSTN基因, 并且在该位点中定点插入了Fat-1基因, 生产出体内既可以高表达Fat-1基因, 提升羊肉中ω-3含量, 又能增加羊肉产量的绒山羊新育种材料 (张驹, 2016) 。角是牛、羊特有的组织结构, 在养殖过程中, 角容易损伤饲养人员, 而人为去角又会违背动物福利且费时费力。Tan等 (2013) 通过TALEN技术以无角安格斯牛1号染色体上重复202 bp为模板, 得到了经基因编辑的荷斯坦牛胎儿成纤维阳性细胞。随后通过体细胞核移植技术成功获得了无角荷斯坦牛, 加快了无角牛新品种的育种进程 (Carlson et al., 2016) 。由于猪体内缺乏功能性的UCP1 (Uncoupling protein 1) 基因, 导致仔猪体温调节不良, 易受寒冷的影响, 2017年中科院动物所赵建国和金万洙团队合作使用CRISPR/Cas9系统, 将外源性小鼠UCP1基因定点插入到到猪内源性UCP1基因位点, 生产出的后代猪提高了产热能力, 降低了新生崽猪由于受冻而死亡的可能, 使抗寒猪的育种迈出了坚实的一步 (Zheng et al., 2017) 。
3.3 应用于家畜乳腺生物反应器研制
动物乳腺生物反应器 (mammary gland bioreactor) 亦称为转基因动物乳腺个体表达系统, 是指利用哺乳动物乳腺特异性启动子调控原件指导目的基因在乳腺中特异性表达, 以期从基因修饰动物乳汁中获取重组蛋白的一种生物反应器 (付玉华等, 2010) , 在牛、羊等家畜动物中具有广泛的应用。
Moghaddassi等 (2014) 年报道了利用TALEN技术结合体细胞核移植技术成功实现了人血清白蛋白 (human serum albumin, hSA) 基因在母牛乳腺中的高表达, 为大规模的生产质量可靠、价格便宜的人源性的血清白蛋白奠定了坚实的基础。Peng等 (2015) 利用最新的CRISPR/Cas9技术, 将猪作为乳腺反应器动物生产出了乳腺中高表达人重组血清白蛋白的转基因猪。Cui等 (2015) 利用TALEN技术以及体细胞核移植技术, 将人乳铁蛋白基因 (human lactoferrin, hLF) 定点插入到奶山羊的β-乳球蛋白 (Beta lactoglobulin) 基因座, 实现了乳铁蛋白在奶山羊乳腺中高表达, 不仅提高了羊奶的营养价值, 而且靶向沉默了β-乳球蛋白基因, 减少了新生儿的乳蛋白过敏反应。
笔者所在实验室朱红梅 (2016) 同样借助TAL-EN这种基因编辑工具, 利用同源重组手段将人乳白蛋白基因 (human lactalbumin, hLA) 定点插入到山羊β-乳球蛋白基因位点。葛恒涛 (2016) 利用TALE切口酶介导的基因打靶技术将口蹄疫病毒 (Foot and mouth disease virus, FMDV) 、猪瘟病毒 (Classical swine fever virus, CSFV) 和PRRSV等具有免疫原性的结构蛋白基因定点整合至山羊β-乳球蛋白基因座, 并通过体细胞核移植技术生产出了乳中可分泌抗病活性蛋白的转基因乳腺反应器奶山羊。此外, 2017年中国农业大学刘国世课题组利用CRISPR/Cas9系统成功在山羊乳腺中过表达参与褪黑素合成的5-羟色胺-N-乙酰基转移酶 (arylalkylamine N-acetyltransferase, AANAT) 基因和乙酰血清素甲基转移酶 (acetylserotonin methyltransferase, ASMT) 基因, 获得高褪黑素含量的功能乳, 为研究生产功能乳提供了研究基础 (Ma et al., 2017) 。
3.4 应用于家畜疾病动物模型的建立
随着新型基因编辑技术的不断发展与完善, 其已广泛的应用于家畜疾病动物模型的建立。PPAR-γ (Peroxisome proliferator-activated receptor-γ) 基因在人类心血管疾病的发病方面具有重要作用。Yang等 (2011) 利用ZFN技术结合体细胞核移植技术生产出了两头PPAR-γ基因敲除猪, 该模型的建立为人类进行心血管疾病基础研究和治疗奠定了基础。低密度脂蛋白受体 (low-density lipoprotein receptor, LD-LR) 是导致家族性高胆固醇血症的重要致病基因, 人类血清低密度脂蛋白升高可导致血液中胆固醇水平升高, 会增加动脉粥样硬化与早发冠心病的风险。Carlson等 (2012) 通过TALEN技术结合体细胞核移植技术生产出了特异性的LDLR基因敲除小型猪, 建立了家族性高胆固醇血症理想的实验动物模型。Hai等 (2014) 利用CRISPR/Cas9系统和显微注射技术成功地对猪的血管性血友病因子基因 (von Willebrand factor, vWF) 进行了靶向沉默, vWF基因突变可使猪体组织出血严重, 出现与血管性血友病相似的动物模型, 为该病的发病遗传基础研究以及治疗研究提供了很好的实验动物模型。
赖良学课题组报道了通过CRISPR/Cas9系统获得成活的酪氨酸酶基因 (tyrosinase, TRY) 敲除猪, TRY基因的敲除致使猪存在典型的白化病表型, 该研究对人类攻克白化病具有重要的应用价值 (Zhou et al., 2015) 。2018年, 该课题组与暨南大学李晓江教授、美国Emory大学李世华教授合作, 同样应用CRISPR/Cas9技术将人源的突变亨廷顿基因 (Huntington, HTT) 基因定点插入到猪HTT基因座内, 经体细胞核移植技术生产出了表达人源性突变型HTT的基因编辑猪, 建立了神经退行性疾病亨廷顿病猪疾病模型 (Yan et al., 2018) 。该模型的建立突出体现了使用大型哺乳动物研究神经退行性疾病及其治疗方法的重要性。帕金森综合症也是老年人中最常见的神经退行性疾病, 体内DJ-1, parkin和PINK1基因中隐性遗传的功能丧失突变可能是导致帕金森综合症的重要病因 (Kitada et al., 1998;Lesage, Brice, 2009;Valente et al., 2004) 。Wang等 (2016) 利用CRISPR/Cas9技术构建出了同时针对巴马小型猪中的三种不同的基因座parkin/DJ-1/PINK1的人帕金森综合症猪模型。
3.5 其他方面的应用
用猪的器官进行人的异体器官移植在克服人体器官严重不足方面具有巨大的应用潜能。临床上, 在猪体内几乎所有组织存在α-1, 3-半乳糖基转移酶 (α-1, 3-galactosyltransferase, GGTA1) 基因所编码的α-1, 3-半乳糖抗原 (α-1, 3-galactose, α-gal) , 此抗原是引起人与猪异种器官移植的超急性排斥反应 (hyperacute rejection, HAR) 的主要因素。Hauschild和Niemann (2011) 利用ZFN技术以及体细胞核移植技术首次实现了在猪体内GGTA1基因的双等位基因敲除, 从而为猪-人的异种器官移植的实现迈出了可喜的一步。除了Gal之外, 单磷酸胞嘧啶-N-乙酰神经氨酸羟化酶 (cytidine monophospho-N-acetylneuraminic acid hydroxylase, CMAH) 基因编码的N-羟乙酰神经氨酸 (N-g1yco1ylneuraminicacid, Neu5Gc) 是另一种主要免疫抗原, Neu5Gc可在GG-TA1基因敲除猪体内广泛表达 (Song et al., 2010) 。Lutz等 (2013) 同样应用ZFN技术和体细胞核移植技术生产出具有GGTA1/CMAH双基因敲除的猪, 并在体外证明了人体血清对GGTA1/CMAH双基因敲除猪的免疫排斥反应相对单基因GGTA1敲除猪来说更小。Martens等 (2017) 利用CRISPR/Cas9技术生产出了同时敲除GGTA1、CMAH以及B4GALNT2 3个基因的猪, 人类血清对该猪异种免疫排斥反应显着降低。这些特异性抗原敲除猪模型的建立, 将为最终培育出适用于临床应用的猪器官奠定了基础。
在新型环保节粮猪的培育过程中, 华南农业大学吴珍芳团队将四个来自微生物的酶类基因bg17A、eg1314、xynB以及eappA插入到猪的基因组, 培育出的转基因猪新育种材料, 饲养过程中氮磷排出显着减少, 同时节约精粮食, 生长速度快, 育肥出栏快, 有望缓解养猪业的环境污染和粮食消耗问题 (Zhang et al., 2018) 。
4 前景与展望
以ZFN、TALEN和CRISPR/Cas为代表的新型基因编辑技术, 已成为非常有价值的分子生物学工具。这三种基因编辑技术大大增强了研究和探索各种模式生物包括家畜动物的能力。研究人员借助这些分子工具, 能够开发以家畜为代表的生物医学疾病模型 (Yan et al., 2018) , 且在家畜的抗病育种 (Gao et al., 2017) 、性状改良 (Wang et al., 2017) 、乳腺生物反应器 (Ma et al., 2017) 等方面具有重要的应用。
相对于ZFN和TALEN技术, CRISPR/Cas系统以其更高的打靶效率和更低的成本, 显示出广泛的应用前景和灵活性。目前, 制约CRISPR/Cas系统发展的主要因素是脱靶效应及PAM序列的制约。科学工作者进行了很多研究来克服这些问题。在降低脱靶效应方面, 有研究发现增加或减少2~3个核苷酸的sgRNA能够减少错配率从而增加靶序列的专一性 (Fu et al., 2014;Kim et al., 2015) 。Doench等 (2016) 创造了新的sgRNA设计规则并建立了人类和小鼠的全基因库, 用基因库设计的sgRNA能够有效减少CRISPR/Cas9系统的脱靶效应。Frock等 (2015) 将Cas9核酸酶改造为Cas9切口酶 (Cas9 nickase, nCas9) , nCas9能够使Cas9蛋白的HNH或Ruv C结构域失活而形成单链断裂 (single-strand breaks, SSBs) 以减少脱靶效应。Qi等 (2013) 把Cas9蛋白改造为Dead Cas9 (dCas9) , dCas9能够使HNH和RuvC两个活性位点均失活, sgRNA介导dCas9与靶序列结合可以抑制基因的起始转录从而降低脱靶脱靶效应。在克服PAM序列的依赖性方面, 有研究发现不同来源的Cas9蛋白可以识别不同的PAM序列, 比如SpCas9识别5'-NGG PAM, NmCas9识别5'-NNNNGATTPAM (Hou et al., 2013) , StCas9识别5′-NNAGAAPAM (Cong et al., 2013) , KKHsaCas9变体可以识别5'-NNGRRTPAM等 (Kleinstiver et al., 2015) 。此外, 2018年东京大学Osamu Nureki教授和麻省理工学院的张锋教授报道了一个SpCas9变体SpCas9-NG, 可以识别5'-NGPAM (Nishimasu et al., 2018) 。Cas9蛋白的多样性扩大了PAM区打靶目标范围, 有助于选择更为合适的靶位点。
CRISPR/Cas技术已经发展为一个强大的基因编辑工具, 随着其新系统如Cpf1、C2c2、BE等的不断涌现, 并呈现多元化发展, 其已成为近几年进行基因组编辑优先选择的技术 (葛陆星等, 2017) 。随着新型基因编辑技术的不断研发拓展与完善, 以及三种技术的联合应用, 在家畜育种、动物模型制作以及生物技术研究中必将得到广泛应用, 推动畜牧业的整体发展。
参考文献:
[]付玉华, 周秀梅, 钱其军.2010.乳腺生物反应器的研究和产业化进展[J].中国畜牧兽医, 37 (8) :45-51. (Fu Y H, Zhou X M, Qian Q J.2010.The current progress of mammary gland bioreactor for research and industry[J]China Animal Husbandry and Veterinary Medicine, 37 (8) :45-51.)
[]葛恒涛.2016.利用山羊乳腺生物反应器制备具有免疫原性病毒结构蛋白的研究[D].博士学位论文, 西北农林科技大学, 导师:张涌, pp.76-94. (Ge H T.2016.Production of immunogenic virus structural proteins by goat mammary gland bioreactor[D]Thesis for ph.D., North West Agriculture and Forestry University, Supervisor:Zhong Y, pp.76-94.)
[]葛陆星, 康健, 董翔宸, 等.2017.CRISPR/Cas9体系的多元化发展和应用[J].农业生物技术学报, 25 (6) :939-953. (Ge L X, KANG J, Dong X C, et al.2017.The diversified development and application of CRISPR/Cas9 system[J].Journal of Agricultural Biotechnology, 25 (6) 939-953.)
[]王少华, 赵盼盼, 刘通, 等.2018.利用CRISPR/Cas9n技术生产抗蓝耳病的基因编辑克隆猪[J].浙江大学学报 (农业与生命科学版) , 44 (2) :157-161. (Wang S H, Zhao P PLiu T, et al.2018.Production of Porcine reproductive and respiratory syndrome virus (PRRSV) -resistant genomeedited cloned pigs using CRISPR/Cas9n system.Journa of Zhejiang University (Agric.&Life Sci.) , 44 (2) :157-161.)
[]张驹.2016.CRISPR/Cas9系统介导羊MSTN基因敲除和定点整合fat-1基因的研究[D].硕士学位论文, 内蒙古大学, 导师:仓明, pp.47-54. (Zhang J.2016.Generation of MSTN gene knock-out and fat-1 knock-in via CRISP-ER/Cas9[D]Thesis for M.S., Inner Mongolia University Supervisor:Cang M, pp.47-54.)
[]朱红梅.2016.TALENs介导的人α-乳白蛋白基因定点敲入β-乳球蛋白位点奶山羊生产[D].博士学位论文, 西北农林科技大学, 导师:靳亚平, pp.71-84. (Zhu HM.2016.TALENs mediated site-specific insertion of human-lactabumin gene into goatβ-lactoglobulin locus[D]Thesis for ph.D., North West Agriculture and Forestry University, Supervisor:Jin Y P, pp.71-84.)
Abudayyeh O O, Gootenberg J S, Konermann S, et al.2016C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector[J].Science, 353 (6299) aaf5573.
Bedell V M, Wang Y, Campbell J M, et al.2012.In vivo genome editing using a high-efficiency TALEN system[J]Nature, 491 (7422) :114-118.
Bibikova M, Beumer K, Trautman J K, et al.2003.Enhancing gene targeting with designed zinc finger nucleases[J]Science, 300 (5620) :764-764.
[]Bonas U, Stall R E, Staskawicz B.1989.Genetic and structural characterization of the avirulence gene avrBs3 from Xanthomonas campestris pv.vesicatoria[J].Molecular&General Genetics Mgg, 218 (1) :127-136.
Burkard C, Lillico S G, Reid E, et al.2017.Precision engineering for PRRSV resistance in pigs:Macrophages from genome edited pigs lacking CD163 SRCR5 domain are fully resistant to both PRRSV genotypes while maintaining biological function[J].PLoS Pathogens, 13 (2) :e1006206.
Calvert J G, Slade D E, Shields S L, et al.2007.CD163 expression confers susceptibility to porcine reproductive and respiratory syndrome viruses[J].Journal of Virology, 81 (14) :7371.
Carlson D F, Fahrenkrug S C, Hackett P B.2012.Targeting DNA With Fingers and TALENs[J].Molecular Therapy Nucleic Acids, 1 (1) :e3.
Carlson D F, Lancto C A, Zang B, et al.2016.Production of hornless dairy cattle from genome-edited cell lines[J].Nature Biotechnology, 34 (5) :479.
Carlson D F, Tan W, Lillico S G, et al.2012.Efficient TAL-EN-mediated gene knockout in livestock[J].Proceedings of the National Academy of Sciences of the USA, 109 (43) :17382-17387.
[]Carroll D.2011.Genome engineering with zinc-finger nucleases[J].Genetics, 188 (4) :773-782.
Cong L, Ran F A, Cox D, et al.2013.Multiplex Genomic Engineering Using CRISPR/Cas Systems[J].Science, 339 (6121) :819-823.
Cui C, Song Y, Liu J, et al.2015.Gene targeting by TALEN-induced homologous recombination in goats directs production ofβ-lactoglobulin-free, high-human lactoferrin milk[J].Scientific Reports, 5:10482.
Delay B D, Corkins M E, Hanania H L, et al.2017.Tissuespecific Gene Inactivation in Xenopus laevis:Knockout of lhx1 in the Kidney with CRISPR/Cas9[J].Genetics, 208 (2) :673-686.
Deltcheva E, Chylinski K, Sharma C M, et al.2011.CRISPRRNA maturation by trans-encoded small RNA and host factor RNase III[J].Nature, 471 (7340) :602-607.
Denecke S, Fusetto R, Batterham P.2017.Describing the role of Drosophila melanogaster ABC transporters in insecticide biology using CRISPR-Cas9 knockouts[J].Insect Biochem Mol Biol, 91 (12) :1-9.
Deng D, Yan C, Pan X, et al.2012.Structural basis for sequence-specific recognition of DNA by TAL effectors[J].Science, 335 (6069) :720-723.
Doench J G, Fusi N, Sullender M, et al.2016.Optimized sgRNA design to maximize activity and minimize offtarget effects of CRISPR-Cas9[J].Nature Biotechnology, 34 (2) :184-191.
Doudna J A, Charpentier E.2014.Genome editing.The new frontier of genome engineering with CRISPR-Cas9[J].Science, 346 (6213) :1258096.
Feng Z, Zhang B, Ding W, et al.2013.Efficient genome editing in plants using a CRISPR/Cas system[J].Cell Research, 23 (10) :1229-1232.
Frock R L, Hu J, Meyers R M, et al.2015.Genome-wide detection of DNA double-stranded breaks induced by engineered nucleases[J].Nature Biotechnology, 33 (2) :179-186.
Fu Y, Foden J A, Khayter C, et al.2013.High-frequency offtarget mutagenesis induced by CRISPR-Cas nucleases in human cells[J].Nature Biotechnology, 31 (9) :822.
Fu Y, Sander J D, Reyon D, et al.2014.Improving CRISPR-Cas nuclease specificity using truncated guide RNAs[J]Nature Biotechnology, 32 (3) :279-284.
Gaj T, Guo J, Kato Y, et al.2012.Targeted gene knockout by direct delivery of ZFN proteins[J].Nature Methods, 9 (8) :805.
Gao Y, Wu H, Wang Y, et al.2017.Single Cas9 nickase induced generation of NRAMP1 knockin cattle with reduced off-target effects[J].Genome Biology, 18 (1) :13.
Gapinske M, Luu A, Winter J, et al.2018.CRISPR-SKIP:Programmable gene splicing with single base editors[J].Genome Biology, 19 (1) :107.
Gaudelli N M, Komor A C, Rees H A, et al.2017.Programmable base editing of A·T to G·C in genomic DNA withou DNA cleavage[J].Nature, 551:464.
Grissa I, Vergnaud G, Pourcel C.2007.The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J].BMC Bioinformatics, 8 (1) :172.
Guo R, Wan Y, Dan X, et al.2016.Generation and evaluation ofMyostatinknock-out rabbits and goats using CRISPRCas9 system[J].Scientific Reports, 6:29855.
Hai T, Teng F, Guo R, et al.2014.One-step generation of knockout pigs by zygote injection of CRISPR/Cas system[J].Cell Research, 24 (3) :372.
[]Hauschild-Quintern J, Petersen B, Cost G J, et al.2013.Gene knockout and knockin by zinc-finger nucleases:Curren status and perspectives[J].Cellular&Molecular Life Sciences Cmls, 70 (16) :2969.
Hauschild J, Niemann H.2011.CORRECTIONS:Efficien generation of a biallelic knockout in pigs using zinc-finger nucleases[J].Proceedings of the National Academy of Sciences of the USA, 108 (29) :12013.
Hinkley S J, Ankoudinova I, Barlow C K, et al.2011.A TALEnuclease architecture for efficient genome editing[J]Nature Biotechnology, 29 (20) :143-148.
[]Hou Z, Zhang Y, Propson N E, et al.2013.Efficient genome engineering in human pluripotent stem cells using Cas9from Neisseria meningitidis[J].Proceedings of the National Academy of Sciences of the USA, 110 (39) 15644-15649.
Hsu P D, Scott D A, Weinstein J A, et al.2013.DNA targeting specificity of RNA-guided Cas9 nucleases[J].Nature Biotechnology, 31 (9) :827.
Ishino Y, Shinagawa H, Makino K, et al.1987.Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J].Journal of Bacteriology, 169 (12) :5429-5433.
Jinek M, Chylinski K, Fonfara I, et al.2012.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science, 337 (6096) :816-821.
[]Karginov F V, Hannon G J.2010.The CRISPR system:Small RNA-guided defense in bacteria and archaea[J].Molecular Cell, 37 (1) :7-19.
Kim D, Bae S, Park J, et al.2015.Digenome-seq:Genomewide profiling of CRISPR-Cas9 off-target effects in human cells[J].Nature Methods, 12 (3) :237-243.
Kim S, Kim J S.2011.Targeted genome engineering via zinc finger nucleases[J].Plant Biotechnology Reports, 5 (1) :9-17.
Kim Y G, Cha J, Chandrasegaran S.1996.Hybrid restriction enzymes:zinc finger fusion to FokI cleavage domain[J].Proceedings of the National Academy of Sciences of the USA, 93 (3) :1156-1160.
Kitada T, Asakawa S, Hattori N, et al.1998.Mutations in the parkin gene cause autosomal recessive juvenile parkinsonism[J].Nature, 392 (6676) :605.
Kleinstiver B P, Prew M S, Tsai S Q, et al.2015.Broadening the targeting range of Staphylococcus aureus CRISPR-Cas9 by modifying PAM recognition[J].Nature Biotechnology, 33 (12) :1293-1298.
[]Klug A.2010.The discovery of zinc fingers and their applications in gene regulation and genome manipulation[J].Annual Review of Biochemistry, 43 (1) :213-231.
Komor A C, Kim Y B, Packer M S, et al.2016.Programmable editing of a target base in genomic DNA without doublestranded DNA cleavage[J].Nature, 533 (7603) :420-424.
Lesage S, Brice A.2009.Parkinson's disease:From monogenic forms to genetic susceptibility factors[J].Human Molecular Genetics, 18 (1) :48-59.
Li T, Huang S, Zhao X, et al.2011.Modularly assembled designer TAL effector nucleases for targeted gene knockout and gene replacement in eukaryotes[J].Nucleic Acids Research, 39 (14) :6315.
Liu X, Wang Y, Guo W, et al.2013.Zinc-finger nickase-mediated insertion of the lysostaphin gene into the beta-casein locus in cloned cows[J].Nature Communications, 4 (Pt 9) :2565.
Liu X, Wang Y, Tian Y, et al.2014.Generation of mastitis resistance in cows by targeting human lysozyme gene toβ-casein locus using zinc-finger nucleases[J].Proceedings of the Royal Society B:Biological Sciences, 281 (1780) :20133368.
Luo J, Song Z, Yu S, et al.2014.Efficient generation of myostatin (MSTN) biallelic mutations in cattle using zinc finger nucleases[J].PLoS One, 9 (4) :e95225.
[]Lutz A J, Li P, Estrada J L, et al.2013.Double knockout pigs deficient in N-glycolylneuraminic acid and Galactoseα-1, 3-Galactose reduce the humoral barrier to xenotransplantation[J].Xenotransplantation, 20 (1) :27-35.
Ma T, Tao J, Yang M, et al.2017.An AANAT/ASMT transgenic animal model constructed with CRISPR/Cas9 system serving as the mammary gland bioreactor to produce melatonin-enrich milk in sheep[J].Journal of Pineal Research, 63 (1) .
Ma X, Zhu Q, Chen Y, et al.2016.CRISPR/Cas9 platforms for genome editing in plants:Developments and applications[J].Molecular Plant, 9 (7) :961-974.
Makarova K, Grishin N, Shabalina S, et al.2006.A putative RNA-interference-based immune system in prokaryotes Computational analysis of the predicted enzymatic machinery, functional analogies with eukaryotic RNAi, and hypothetical mechanisms of action[J].Biology Direct1:7.
[]Mani M, Kandavelou K, Durai S, et al.2005.Design, engineering, and characterization of zinc finger nucleases[J]Biochemical&Biophysical Research Communications335 (2) :447-457.
Martens G R, Reyes L M, Butler J R, et al.2017.Humoral reactivity of renal transplant-waitlisted patients to cells from GGTA1/CMAH/B4GalNT2, and SLA class Iknockout pigs[J].Transplantation, 101 (4) :e86.
Mcpherron A C, Lee S J.1997.Double muscling in cattle due to mutations in the myostatin gene[J].Proceedings of the National Academy of Sciences of the USA, 94 (23) 12457-12461.
Moghaddassi S, Eyestone W, Bishop C E.2014.TALEN-mediated modification of the bovine genome for largescale production of human serum albumin[J].PLoSOne, 9 (9) :e89631.
[]Mojica F J, Díez-Villase?or C, Soria E, et al.2000.Biologica significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria[J]Molecular Microbiology, 36 (1) :244-246.
Mojica F J, Montoliu L.2016.On the Origin of CRISPR-Cas Technology:From Prokaryotes to Mammals[J].Trends in Microbiology, 24 (10) :811-820.
Moscou M J, Bogdanove A J.2009.A simple cipher governs DNA recognition by TAL effectors[J].Science, 326 (5959) :1501-1501.
Nakasuji T, Ogonuki N, Chiba T, et al.2017.Complementary critical functions of Zfy1 and Zfy2 in mouse spermatogenesis and reproduction[J].PLoS genetics, 13 (1) :e1006578.
Nakasuji T, Ogonuki N, Chiba T, et al.2017.Complementary critical functions of Zfy1 and Zfy2 in mouse spermatogenesis and reproduction[J].Plos Genetics, 13 (1) :e1006578.
Nishimasu H, Shi X, Ishiguro S, et al.2018.Engineered CRISPR-Cas9 nuclease with expanded targeting space[J].Science.
Osborn M J, Starker C G, Mcelroy A N, et al.2013.TALEN-based gene correction for epidermolysis bullosa[J].Molecular Therapy, 21 (6) :1151-1159.
Palpant N J, Dudzinski D.2013.Zinc finger nucleases:Looking toward translation[J].Gene Therapy, 20 (2) :121-127.
Pattanayak V, Ramirez C L, Joung J K, et al.2011.Revealing off-target cleavage specificities of zinc-finger nucleases by in vitro selection[J].Nature Methods, 8 (9) :765-770.
Peng J, Wang Y, Jiang J, et al.2015.Production of Human Albumin in Pigs Through CRISPR/Cas9-Mediated Knockin of Human cDNA into Swine Albumin ocus in the Zygotes[J].Scientific Reports, 5:16705.
[]Qi L S, Larson M H, Gilbert L A, et al.2013.Repurposing CRISPR as an RNA-Guided Platform for Sequence-Specific Control of Gene Expression[J].Cell, 152 (5) :1173-1183.
Radecke S, Radecke F, Cathomen T, et al.2010.Zinc-finger Nuclease-induced Gene Repair With Oligodeoxynucleotides:Wanted and Unwanted Target Locus Modifications[J].Molecular Therapy, 18 (4) :743-753.
Ran F A, Cong L, Yan W X, et al.2015.In vivo genome editing using Staphylococcus aureus Cas9[J].Nature, 520 (7546) :186-191.
Richt J A, Kasinathan P, Hamir A N, et al.2007.Production of cattle lacking prion protein[J].Nature Biotechnology, 25 (1) :132-138.
Roper J, Tammela T, Akkad A, et al.2018.Colonoscopybased colorectal cancer modeling in mice with CRISPR-Cas9 genome editing and organoid transplantation[J].Nature Protocols, 13 (2) :217-234.
Ryder P, Mchale M, Fort A, et al.2017.Generation of stable nulliplex autopolyploid lines of Arabidopsis thaliana using CRISPR/Cas9 genome editing[J].Plant Cell Reports, 36 (6) :1005-1008.
Shan Q, Wang Y, Li J, et al.2013.Targeted genome modification of crop plants using a CRISPR-Cas system[J].Nature Biotechnology, 31 (8) :686-688.
Shipman S L, Nivala J, Macklis J D, et al.2017.CRISPR-Cas encoding of a digital movie into the genomes of a population of living bacteria[J].Nature, 547 (7663) .
Song K H, Kang Y J, Jin U H, et al.2010.Cloning and functional characterization of pig CMP-N-acetylneuraminic acid hydroxylase for the synthesis of N-glycolylneuraminic acid as the xenoantigenic determinant in pig-human xenotransplantation[J].Biochemical Journal, 427 (1) :179-188.
Sorek R, Kunin V, Hugenholtz P.2008.CRISPR--a widespread system that provides acquired resistance agains phages in bacteria and archaea[J].Nature Reviews Microbiology, 6 (3) :181-186.
[]Tan W, Carlson D F, Lancto C A, et al.2013.Efficient nonmeiotic allele introgression in livestock using custom endonucleases[J].Proceedings of the National Academy of Sciences of the USA, 110 (41) :16526-16531.
[]Ul A Q, Chung J Y, Kim Y H.2015.Current and future delivery systems for engineered nucleases:ZFN, TALEN and RGEN[J].Journal of Controlled Release, 205:120-127.
Urnov F D, Rebar E J, Holmes M C, et al.2010.Genome editing with engineered zinc finger nucleases[J].Nature Reviews Genetics, 11 (9) :636-646.
Valente E M, Abousleiman P M, Caputo V, et al.2004.Hereditary early-onset Parkinson's disease caused by mutations in PINK1[J].Science, 304 (5674) :1158-1160.
Verma N, Zhu Z, Huangfu D.2017.CRISPR/Cas-Mediated Knockin in Human Pluripotent Stem Cells[J].Methods in Molecular Biology, 1513:119-140.
Wang K, Tang X, Xie Z, et al.2017.CRISPR/Cas9-mediated knockout of myostatin in Chinese indigenous Erhualian pigs[J].Transgenic Research, 26 (6) :1-7.
Wang X, Cao C, Huang J, et al.2016.One-step generation of triple gene-targeted pigs using CRISPR/Cas9 system[J]Scientific Reports, 6:20620.
Wang X, Yu H, Lei A, et al.2015.Generation of gene-modified goats targeting MSTN and FGF5 via zygote injection of CRISPR/Cas9 system[J].Scientific Reports, 513878.
Wang X L, Niu Y Y, Zhou J K, et al.2016.Multiplex gene editing via CRISPR/Cas9 exhibits desirable muscle hypertrophy without detectable off-target effects in sheep[J]Scientific Reports, 6:32271.
Whitworth K M, Rowland R R, Ewen C L, et al.2015.Geneedited pigs are protected from porcine reproductive and respiratory syndrome virus[J].Nature Biotechnology, 34 (1) :20.
Wu H, Wang Y, Zhang Y, et al.2015.TALE nickase-mediated SP110 knockin endows cattle with increased resistance to tuberculosis[J].Proceedings of the National Academy of Sciences of the USA, 112 (13) :E1530.
Wyman C, Kanaar R.2006.DNA double-strand break repair:All's well that ends well[J].Annual Review of Genetics, 40 (1) :363.
Xie Z X, Mitchell L A, Liu H M, et al.2018.Rapid and Efficient CRISPR/Cas9-Based Mating-Type Switching of Saccharomyces cerevisiae[J].G3 Genesgenetics, 8 (1) :173-183.
Yan S, Tu Z, Liu Z, et al.2018.A huntingtin knockin pig model recapitulates features of selective neurodegeneration in Huntington's disease[J].Cell, 173 (4) .
Yang D, Yang H, Li W, et al.2011.Generation of PPARγmono-allelic knockout pigs via zinc-finger nucleases and nuclear transfer cloning[J].Cell Research, 21 (6) :979-982.
Yao X, Liu Z, Wang X, et al.2018.Generation of knock-in cynomolgus monkey via CRISPR/Cas9 editing[J].Cell Research, 28 (3) :379-382.
Yin X, Biswal A K, Dionora J, et al.2017.CRISPR-Cas9 and CRISPR-Cpf1 mediated targeting of a stomatal developmental gene EPFL9 in rice[J].Plant Cell Reports, 36 (5) :745-757.
Yu B, Lu R, Yuan Y, et al.2016.Efficient TALEN-mediated myostatin gene editing in goats[J].Bmc Developmental Biology, 16 (1) :26.
[]Zetsche B, Gootenberg J S, Abudayyeh O O, et al.2015.Cpf1is a single RNA-guided endonuclease of a Class 2 CRIS-PR-Cas system[J].Cell, 163 (3) :759-771.
Zhang X, Li Z, Yang H, et al.2018.Novel transgenic pigs with enhanced growth and reduced environmental impact[J].Elife, 7.
Zhang Y, Qin W, Lu X, et al.2017.Programmable base editing of zebrafish genome using a modified CRISPR-Cas9 system[J].Nature Communications, 8 (1) :118.
Zheng Q, Lin J, Huang J, et al.2017.Reconstitution of using CRISPR/Cas9 in the white adipose tissue of pigs decreases fat deposition and improves thermogenic capacity[J].Proceedings of the National Academy of Sciences of the USA, 114 (45) :E9474.
Zhou X, Xin J, Fan N, et al.2015.Generation of CRISPR/Cas9-mediated gene-targeted pigs via somatic cell nuclear transfer[J].Cellular&Molecular Life Sciences, 72 (6) :1175-1184.





