
引言
【研究意义】血液中的血糖(glucose,GLU)循环全身,对人体具有重要的生理功能,是反映机体生理状况的一个重要指标。空腹血糖浓度(fasting serumglucose, FSG)是糖尿病临床诊断的常规指标,也是糖尿病诊断后 10 年内因心血管疾病死亡的独立预警指标。糖尿病病人的空腹血糖浓度持续偏高,波动较小,是一种营养代谢性疾病。有研究表明,空腹血糖浓度是可遗传的,遗传力为0.62 ± 0.08(P<0.01),属于高遗传力性状。糖基化血清蛋白(glycosylatedserum proteins,GSP)是一个比血糖更稳定、更能反映机体 2—3 周内平均血糖水平的指标。猪作为模式动物,与人类具有相似的代谢功能、心血管系统、成比例的器官大小等,已经成为研究人类肥胖、糖尿病及并发症等疾病的最佳模式动物。研究影响猪 GLU和 GSP 的染色体位点及候选基因定位,不仅对猪代谢生理功能的遗传解析具有重要意义,而且对人类相关疾病的临床研究具有参考价值。【前人研究进展】近年来,已定位了多个影响 GLU 和 GSP 的 QTL。
Cai 等在对西班牙裔儿童研究中发现,影响空腹血糖浓度的 QTL 定位在 13 号染色体的 85—99 cM处;An 等在人 19 号染色体的 88cM 处定位到了显着影响空腹血糖浓度的 QTL,在 7 号染色体的 163cM处定位到了染色体可显着影响空腹血糖浓度的 QTL;Kobayashi 等在由 SM×SMXA-5 构建的 F2小鼠中,在 8 号染色体上定位到了影响空腹血糖浓度的 QTL。
人和鼠的血糖性状 QTL 定位结果具有很高的一致性。在对猪的研究中,Yoo 等利用长白×韩国本地猪种 F2群体,在 SSC1 和 SSC8 上定位到染色体可显着影响 GLU 的 QTL;Désautés 等利用梅山猪和大白猪构建的 F2群体在 SSC1、3、5、8 四条染色体上定位到了影响 GLU 浓度的 QTL;陈蓉蓉等在白色杜洛克×二花脸资源家系 SSC14 的 68 cM 处定位到了 1 个 5%基因组显着影响 GLU 的 QTL。由猪 QTL数据库可知,影响 GSP 的 QTL 定位较少,目前只有在SSC4的54 cM处发现一个染色体显着水平QTL。
【本研究切入点】目前,尚未见利用 Illumina porcine60K SNP 芯片进行全基因组关联分析定位影响猪GLU 和 GSP 染色体位点的研究报道。【拟解决的关键问题】本研究利用猪 60K SNP 芯片,通过全基因组关联分析来定位影响猪GLU和GSP的染色体位点,为进一步鉴别影响猪 GLU 和 GSP 的主效基因和因果突变位点提供研究基础,为人类低血糖症和糖尿病的遗传学研究提供参考。
1 材料与方法
本试验于 2011 年 3 月至 2013 年 9 月在江西农业大学动物生物技术国家重点实验室培育基地进行。
1.1 试验动物
试验猪为中国培育猪种苏太猪和江西农业大学动物生物技术国家重点实验室培育基地构建的白色杜洛克×二花脸 F2资源家系。F2资源家系以 2 头白色杜洛克公猪和 17 头二花脸母猪为 F0始祖,杂交产生F1代。随机选择 9 头 F1公猪和 59 头 F1母猪,分六批次共繁育 1912 头 F2个体,其中 760 头 F2个体(411 头公猪,349 头母猪)屠宰时采集血液,分离血清用于 GLU 和 GSP 测定。所有 F2仔猪 46 d 断奶,公猪 90 d 阉割。435 头苏太猪(228 头公猪和 207 头母猪)购自苏州市苏太育种中心,18 d 阉割,28 d断奶。两品种猪按统一常规方法饲养,采食相同饲料,自由采食和饮水。(240±5)日龄禁食禁水 24 h 后进行屠宰取样。
1.2 血清 GLU 和 GSP 含量测定
血液样品在室温下静置 5 h,4℃ 3 000 r/min 离心20 min,分离血清保存于-80℃待测。血清样品送至南昌 大 学 第 一 附 属 医 院 使 用 全 自 动 生 化 分 析 仪(AU5421,美国贝克曼库尔特)测定 GLU 和 GSP 含量。
1.3 DNA 提取及 60K SNP 芯片判型采集
0.5—1.0 g 猪耳组织,按照常规酚/氯仿法提取基因组 DNA。用 NANODROP 1000 核酸蛋白分析仪(Thermo Scientific,USA)测定 DNA 浓度和质量,A260/280的比值在 1.8—2.0 判定合格,将浓度统一稀释至 20 ng·μL-1,-20℃冰箱保存。
DNA 样品利用 Illumina Bead Array SNP 基因分型平台进行猪 60K SNP 芯片分型(北京怡美通德科技发展有限公司)。利用 PLINK 软件对 SNP 进行质控,将检测率≥95.0%、次等位基因频率(minor allelefrequency)≥0.05 且符合家系分离规律的 SNP 标记用于后续的统计分析。
1.4 统计分析
采用广义线性混合模型判定各个 SNP 与表型性状之间关联性,分析模型为:y=μ+Xb+Sc+Zα+e。其中,μ 为性状平均值,b 为性别和批次的固定效应向量,c 为其余的微效应,α 为随机加性遗传效应向量,e 为残差,X、S 和 Z 是对固定效应和随机效应的关联矩阵,y为所测得的表型值,利用R语言中的GenABEL软件包进行 GWAS 分析。整合 F2资源家系和苏太猪共有的 34495 个通过质量控制的 SNP 进行 GLU 和GSP 性状的 Meta GWAS 分析,当自由度为 3 时,根据每一个 SNP 位点在每个群体中的卡方值(x2)计算合并的新的 x2值,利用 Bonferroni 校正多重检验确定显着性阈值,基因组显着水平阈值为 0.05/有效SNP 数量,染色体显着水平阈值为 1/有效 SNP 位点数量。
1.5 位置候选
基因搜寻利用 Ensemb或 NCBI猪参考基因组数据库中搜寻关联性显着的 SNP 所在区域的已知基因信息,根据基因注释情况分析可能的位置候选基因。
2 结果
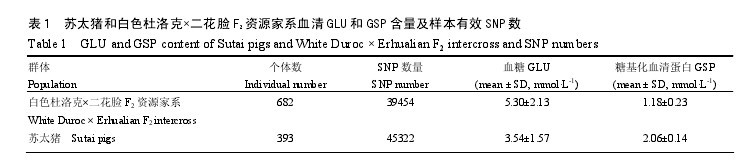
2.1 血清 GLU 和 GSP 含量测定苏太猪和 F2资源家系血清 GLU 和 GSP 含量测定结果见表 1。两个群体通过质控的有效表型数据个体数分别为 682 头和 393 头;从表中可以看出,F2资源家系血清平均 GLU 浓度高于苏太猪,平均 GSP 浓度低于苏太猪。【表1】
2.2 全基因组关联分析
GLU 和 GSP 两性状的全基因组关联分析结果见表 2。本试验共检测到 3 个 SNP 位点与 GSP 相关,2个 SNP 位点与 GLU 相关,这些 SNP 与表型的关联性只达到染色体显着水平。【表2】
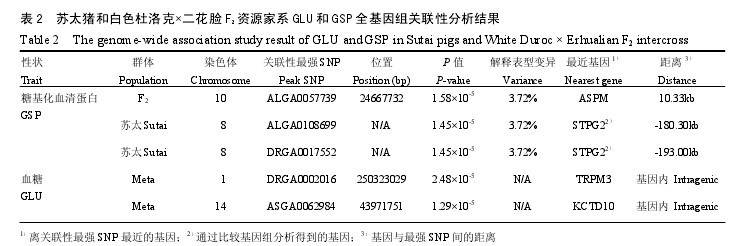
在白色杜洛克×二花脸 F2资源群体中,10 号染色体上的 SNP ALGA0057739与血清 GSP含量的关联性达染色体显着水平(P=1.58×10-5,图 1),该 SNP 解释表型变异为 3.72%,位于染色体 24.67Mb 处,ASPM 基因距离该 SNP 最近(10.33kb)。
在苏太猪群体中,检测到两个与 GSP 达染色体显着水平的 SNP(ALGA0108699 和 DRGA0017552,P=1.45× 10-5,图 2),SNP 所解释的表型变异均为3.72%。使用猪 10.2 版本参考基因组序列,这两个 SNP均无法定位到具体的染色体位置(表 2)。通过人、猪比较基因组分析发现,这两个 SNP 均位于 SSC8,处于 STPG2 基因 3’下游 180.3—193.0kb 处。将两个试验猪群体合并,对 GSP 性状进行 Meta 分析,并未发现与 GSP 显着相关的 SNP 位点(图 3)。
在白色杜洛克×二花脸 F2资源家系和苏太猪两个群体中分别进行 GWAS 分析,并未定位到与血清GLU 含量显着相关的 SNP。两个群体进行 Meta 分析时,1 号染色体 250.32Mb 处的 SNP DRGA0002016 与GLU 的关联性达染色体显着水平(P = 2.48×10-5),SNP位于TRPM3 基因内;在14 号染色体43.97Mb 处也分析得到一个达染色体显着水平的SNP(ASGA0062984,P =1.29×10-5),SNP 位于 KCTD10 基因内(图 4)。
3 讨论
本试验在苏太猪和 F2资源群体中共检测到 3 个SNP 与血清 GSP 含量显着相关,但定位结果与 Chen等在相同 F2资源群体中基于 194 个微卫星标记的QTL 定位结果不一致。QTL 定位分析与全基因组关联分析是不同的,首先 QTL 定位分析是基于 QTL 的两个等位基因在 F0始祖中固定这个假设基础上进行的,全基因组关联分析无此假设,因此即使 QTL 在始祖中分离,GWAS 也能检测到该显着性位点;在 GWAS分析中,仅考虑了加性效应,而在 QTL 定位中同时考虑了加性和显性效应;GWAS 分析中使用的 SNP 标记的密度远大于 194 个微卫星标记。在苏太猪和 F2资源群体中检测到的与 GSP 显着关联的 SNP 不一致,表明影响 GSP 的染色体位点存在群体异质性。本试验在苏太猪和 F2资源群体中均未检测到与 GLU 显着相关的 SNP,可能是群体样本数量不足导致。本试验获得的关联性显着相关的 SNP 其解释的表型变异<5%,要检测到效应较小的染色体位点需要较大的群体规模。另外与 GSP 相比,血清中 GLU 的含量更易受到环境等因素的影响。
对于某些染色体位点,如果对不同群体同一性状具有相同的作用,通过合并不同群体进行 Meta 分析可以起到增加群体样本规模的作用,有可能发现新的显着性位点。本试验合并两个群体进行两个性状的 Meta 分析,结果在 SSC1 和 SSC14 定位到了 2 个与 GLU 显着关联的 SNP,其中 SSC14 上显着关联的SNP 位于 Chen 等报道的影响 GLU 的 QTL 置信区间内,SSC1 上定位到显着关联的SNP 与Désautés 等报道的影响 GLU 的 QTL 处于同一染色体区域。Meta分析未发现新的与 GSP 显着相关的 SNP。
利用猪参考基因组信息(10.2 版),搜寻了离关联性最强 SNP 最近的基因。对于 SSC10,24.67Mb 处显着影响 GSP 的 SNP,在 Ensembl 上搜索到 ASPM基因离其最近(10.33kb),在 GeneCards搜索这个位置候选基因的功能,发现ASPM 可通过调节 Wnt 信号通路而影响大脑发育,Wnt 信号通路与糖尿病密切相关,因此 ASPM 基因是该染色体区域的重要候选基因。SSC1 上与 GLU显着关联的 SNP DRGA0002016 位于 TRPM3 基因内,TRPM3 与钙离子通道介导的钙离子进入细胞有关,在钙离子介导的非选择性通道中广泛表达,TRPM (melastatin)与血糖稳态和代谢综合征相关。
Wagner 等研究小鼠胰腺 β 细胞信号转导时发现,TRPM3 基因介导孕烯醇酮磷酸化刺激胰岛素分泌,TRPM3 基因调节胰岛素的分泌和生物合成。因此,TRPM3 是与 GLU 相关的一个重要候选基因。对于SSC14,43.97Mb 上与 GLU 显着关联的 ASGA0062984位于 KCTD10 基因内,KCTD10 基因与 MVK、MMAB基因紧密相邻,已证实含有该基因的基因调控网络与糖尿病、肥胖、动脉粥样硬化相关,是一个重要的候选基因。离 SSC8 与 GSP 显着相关的 SNPALGA0108699 和 DRGA0017552 最近的 STPG2 基因,在人和其他哺乳动物中研究较少,目前还未见与糖代谢相关的报道。
4 结论
试验测定了 760 头白色杜洛克×二花脸资源家系F2个体和 435 头苏太猪的血清 GSP 和 GLU 的浓度,运用全基因组关联分析得到 5 个与 GLU 和 GSP 显着相关的 SNP,这些 SNP 位于 4 不同染色体区域,解释的表型变异都为 3.72%。通过搜寻显着相关 SNP 区域内的候选基因及其功能注释,发现 ASPM、TRPM3、KCTD10 和 STPG2 基因为影响猪血清 GLU 和 GSP 的重要候选基因。





