摘 要: 基因编辑是一项能够在生物体基因组水平上实现对DNA序列精确定向修饰的新技术。该技术主要利用序列特异性核酸酶靶向识别切割基因组上目标位点,造成DNA双链断裂(DSBs),进一步诱发非同源末端连接(NHEJ)和同源性重组(HR)2种修复机制对断裂的DNA双链进行修复,实现修复位点碱基的插入、缺失和替换,从而达到对靶基因精确编辑的目的。本研究介绍了基因编辑技术的类型、作用原理及该技术在植物领域中的应用,并对基因编辑技术在药用植物功能基因组学和遗传改良中的应用前景进行展望。
关键词: 基因编辑; 药用植物; 应用; 展望;
Abstract: Gene editing is a new technology that enables precise targeted modification of DNA sequences at the genome level of an organism. The technology mainly uses sequence-specific nuclease to target and cleave the target sites on the genome, resulting in DNA double-strand breaks (DSBs), further inducing non-homologous end joining (NHEJ) and homologous recombination (HR) repair mechanisms. The cleavage of the DNA double strand is repaired, and the insertion, deletion and substitution of the base of the repair site are achieved, thereby achieving the purpose of accurately editing the target gene. This paper introduces the type and mechanism of gene editing technology and its application in plant field, and prospects the application of gene editing technology in functional genomics and genetic improvement of medicinal plants.
Keyword: gene editing; medicinal plants; application; prospect;
基因编辑技术ZFN、TALEN、CRISPR/Cas等,是近年来兴起的能够对特定基因位点实现靶向精确编辑的一种先进基因修饰技术。相较于传统的转基因技术而言,基因编辑不仅能够对目标基因进行精确编辑,而且受体生物基因组中无外源DNA片段的引入,不存在食品安全风险,可直接应用于生产[1],尤其是具有操作更简单、编辑更高效、价格更低廉等优点的CRISPR/Cas9,已被大量应用于植物基因组功能和作物遗传改良等相关研究。药用植物的栽培和使用在我国有着悠久的历史,其目标产品中药材在保障人类健康方面发挥着至关重要的作用。但是药用植物在生产上存在许多急需解决的问题,如药用植物种类繁多,且多为野生种质资源,驯化改良周期长;药用植物产量和质量的不稳定性;病虫害种类多、防治难度大;杂草危害严重,药用植物对除草剂敏感等。基因编辑技术在提升药用植物生产水平、改善药用植物品质、产量和抗性等方面具有较大的应用潜力。本研究介绍了基因编辑技术的类型和作用原理及在植物不同领域中的应用,进一步展望该技术在药用植物中的应用前景。

1、 基因编辑技术的类型及作用原理
基因编辑技术可以在生物体基因组水平上对目标基因进行编辑,实现DNA修复位点碱基的敲除、插入和替换等,以达到定向修饰靶基因序列的目的。目前基因编辑技术根据核酸酶的不同主要分为3类:锌指核酸内切酶(ZFN)[2]、类转录激活因子效应物核酸酶(TALEN)[3]和成簇的规律间隔的短回文重复序列(CRISPR)[4]。3者对靶基因的编辑方式类似,均是在目标基因位点通过序列特异性核酸酶(SSN)识别切割靶DNA后造成DNA 双链断裂(DSBs),通过细胞内非同源末端连接(NHEJ)和同源性重组(HR)2种机制进行修复,造成编辑位点碱基的插入、缺失、替换等(见图1),从而实现基因靶位点的精确修饰。其中ZFN和TALEN对靶DNA的识别利用DNA-蛋白质识别原理[5],而CRISPR/Cas9利用RNA-DNA识别原理。
图1 3种基因编辑技术及DSB修复机制示意图
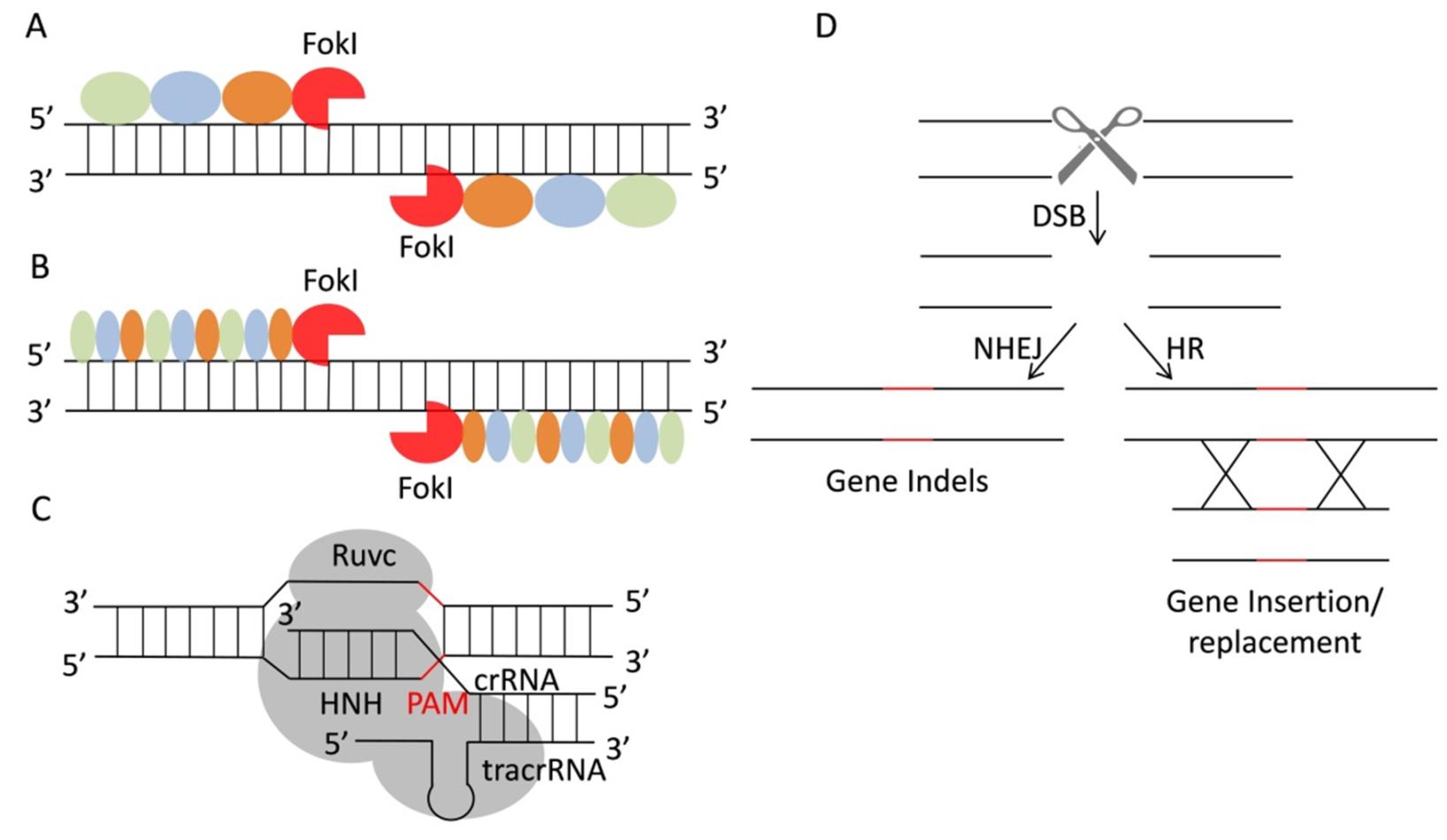
注:A为ZFN基因编辑示意图;B为TALEN基因编辑示意图;C为CRISPR/Cas9基因编辑示意图;D为DSB修复机制
第I代基因编辑技术ZFN是由能够特异识别结合DNA片段的ZFP结构域和对靶DNA片段进行切割的非特异性Fok I核酸内切酶构成。ZFP结构域由3 ~ 6个Cys2His2锌指蛋白组成,每个锌指单元决定了对DNA单链上3个连续核苷酸的特异性识别,因此通过串联多个锌指单元形成ZFP结构域以实现对特定靶序列的识别[6]。基于切割域Fok I核酸内切酶以二聚体形式发挥内切酶作用的特性[7],需在靶位点两端设计正反向排列且间隔5 ~ 7 bp的1对ZFN,从而实现Fok I 结构域在2条靶序列间隔位点的切割,造成DSB[8,9];但是,ZFN设计复杂、成本高、脱靶率高,产生细胞毒性等缺陷限制了该技术的广泛应用。
TALEN是继ZFN之后的第II代基因编辑技术,由特异性识别结合靶DNA的TALE蛋白和Fok I内切酶结合而成(见图1),其中Fok I内切酶和ZFN中的类似,TALE蛋白由N端转录信号、转录激活结构域和C端核定位信号3部分组成。在TALE蛋白的转录激活结构域中存在一段由34个氨基酸组成的非完美重复序列,其中第12、13位重复可变的双氨基酸残基(RVD)决定了1个重复单元对不同核苷酸的特异性识别[10]。不同氨基酸组合形式的RVD与核苷酸的识别存在对应关系,一般为天冬氨酸-异亮氨酸(NI)识别腺嘌呤(A)、天冬氨酸-甘氨酸(NG)识别胸腺嘧啶(T)、天冬氨酸-组氨酸(NH)识别鸟嘌呤(G)、天冬氨酸-天冬氨酸(NN)识别A或G、组氨酸-天冬氨酸(HD)识别胞嘧啶(C)、天冬氨酸-丝氨酸(NS)识别A、T、C或G任1个,据此针对靶序列设计成对的含有18 ~ 20个重复单元的TALEN对靶基因进行定点编辑[10,11]。虽然TALEN在特异性识别切割靶基因方面有了很大的改善,但是其成本高、载体过大、构建复杂等不足同样制约了TALEN技术在植物中的广泛应用。
CRISPR/Cas系统是在细菌和古细菌中发现的一种适应性免疫系统,由5'端的反式激活RNA基因(trans-activating RNA,tracrRNA)、CRISPR位点附近的成簇关联基因(CRISPR associated, Cas)以及CRISPR基因座组成[12]。TracrRNA在促使pre-crRNA形成成熟的小crRNA[13]及协助crRNA-Cas复合体实现精确定位方面发挥重要作用[14]。CRISPR基因座由启动转录的前导序列(leader sequence)、不连续的高度保守的重复序列(repeat)和长度相似的间隔序列(spacer)相间排列而成[15]。其中,前导序列主要负责启动CRISPR的转录;重复序列一般由约21 ~ 48个碱基对组成,其中包含1个由5 ~ 7个碱基对组成的回文结构,其转录物能形成稳定的二级发卡结构[16];间隔序列一般约有26 ~ 72个碱基对组成,是宿主细胞特异性识别外源基因入侵的主要来源[15]。Cas蛋白包含了HNH和RuvC-like2个结构域,其中HNH结构域能够切割与crRNA互补的DNA链,RuvC-like结构域使非互补的DNA链断裂[14]。TracrRNA、crRNA和Cas蛋白3者结合形成TracrRNA-crRNA-Cas三元复合结构,进一步对靶位点3'端PAM邻近的20个核苷酸序列识别切割形成DSB。
尽管CRISPR/Cas9系统是目前应用较为广泛的基因编辑技术,但crRNA的20nt间隔特异识别序列能够忍受一定程度的靶标碱基错配,从而产生较高的脱靶诱导,而且其特异性识别受PAM的制约,限制了该技术的应用范围[17]。在CRISPR/Cas9系统的前提下衍生出CRISPR/Cas9n[18]、CRISPR/dCas9[19]技术实现了脱靶率的降低。2015年9月张峰团队发现的CRISPR/Cas系统的II类V型新成员Cpf1,其能够识别富含胸腺嘧啶(T)的PAM[20],进一步提高了CRISPR/Cas系统的特异性并扩大其识别应用范围,此外Cpf1蛋白与crRNA矩阵相结合,还能够实现对基因组多基因的同时编辑[21]。
2 、基因编辑技术在植物领域中的应用
基因编辑技术通过基因敲除、插入及基因替换等手段,实现对目标基因的定向编辑并获得相应的突变体。近年来,基因编辑技术已经在模式植物拟南芥中得到深入的研究。除此之外,基因编辑技术在大田作物、园艺作物及药用植物中的应用也日趋广泛,其相关研究成果也越来越多。
2.1、 基因编辑技术在大田作物中的应用
早期的TALEN基因编辑技术已经在水稻Oryza sativa中实现对个别基因的定向编辑。Li等[22]通过TALEN基因编辑技术对水稻白叶枯病易感基因OsSWEET14定向编辑,获得具有白叶枯抗性的水稻新品种。姜明君等[23]利用TALEN技术对水稻中已知的苯达松抗性基因(CYP81A6)定向敲除获得苯达松易感性的水稻品种。随着CRISPR/Cas9基因编辑技术研究热潮的兴起,该技术也成功用于水稻的性状和品种改良等相关研究。利用CRISPR/Cas技术对水稻中乙酰乳酸合成酶基因(ALS)[24]、乙烯反应因子基因(OsERF922)[25]、5-烯醇丙酮莽草酸-3-磷酸合成酶基因(OsEPSPS)[26]的靶向修饰,分别获得了除草剂、稻瘟病和草甘膦抗性的水稻品种。利用CRISPR/Cas9对水稻中编码甜菜碱醛脱氢酶的香味控制基因(Badh2)[27]和编码吲哚乙酸-葡萄糖水解酶的千粒重控制基因(tgw6)[28]进行编辑,获得的后代水稻突变体的香味和千粒重都分别得到了显着增加。
Shukla等[29]利用最早的基因编辑技术ZFN靶向编辑玉米Zea mays的IPK1基因,从而增加了玉米对除草剂的耐受性。Char等[30]利用TALEN对玉米的glossy2(gl2)基因进行靶向诱变使该基因失活,并进一步验证了突变体后代中3个等位基因的功能。Shi等[31]利用CRISPR/Cas9基因编辑技术获得了玉米乙烯反应负调控基因(ARGOS8)突变体,其抗旱性得到了显着增强,在干旱条件下改善了玉米籽粒的产量。
除此以外,基因编辑技术在大豆Glycine max[32]、小麦Triticum aestivum[33]、大麦Hordeum vulgare[34]、棉花Gossypium hirsutum[35]等大田作物中也实现了对特异目标位置靶基因的精确编辑,并获得了相应的基因突变体,进一步实现对大田作物性状和品种的改良并获得抗性新品种。
2.2 、基因编辑技术在园艺作物中的应用
番茄Solanum lycopersicum是重要的园艺作物代表之一,基因编辑技术在番茄中的应用也较为广泛。2014年Lor等[36]利用TALEN靶向诱变番茄的GA信号传导负调节剂基因(PROCERA),其获得的pro_7突变体表现出长节间和平滑叶缘的特征。2015年?ermák等[37]基于TALEN基因编辑技术靶向修饰番茄中控制花青素苷生物合成的内源基因ANT1,在ANT1基因的上游插入花椰菜花叶病毒(CaMV)35s启动子,导致番茄组织中的色素过表达和异位积累,从而获得高花青素含量的紫色番茄新种质。近年来,CRISPR/Cas9基因编辑技术也被应用于番茄基因功能的相关研究,Ito等[38]利用CRISPR/Cas9基因编辑技术靶向诱变番茄中能够编码调节果实成熟的MADS-box转录因子的RIN基因,获得的RIN蛋白缺陷突变体产生不完全成熟的番茄果实,且突变体番茄果实红色色素积累显着低于野生型。Ueta等[39]使用CRISPR/Cas9系统成功将1种控制单性结实的SlIAA9基因引入番茄中,所获得的番茄T0代突变体的突变率达100%,且再生突变体表现出番茄单性结实的形态特征简单叶表型和形成无籽果实。近期,北京农林科学院蔬菜中心等单位利用CRISPR/Cas9技术对3个红色果皮番茄杂交种的父母本分别进行基因编辑,获得没有外源基因片段的粉色果皮自交系,重新进行组配,获得的杂种一代果实均为粉色果皮[40],有效地实现了基因编辑技术对番茄新品种的定向创制。
除了番茄,基因编辑技术也在其他园艺作物中得到应用。在马铃薯S. tuberosum中以选择组成型表达的泛素7基因(Ubi7)的5'UTR内的内含子区域作为靶位点,利用TALEN技术将在马铃薯基因组中获得的天然无启动子的除草剂抗性标记和目的转基因的盒插入该靶位点,内源性Ubi7启动子将驱动无启动子的除草剂抗性标记基因表达,从而获得具有除草剂抗性的马铃薯[41]。Kannan等[42]利用TALEN技术对甘蔗Saccharum officinarum中的木质素生物合成基因咖啡酸-O-甲基转移酶(COMT)进行编辑,进而提高甘蔗中的糖化效率。Tian等[43]利用CRISPR/Cas9技术对西瓜中的八氢番茄红素去饱和酶基因(CiPDS)定向诱变,并获得白化表型的突变体植株,实现了基因编辑技术在西瓜中的应用。Klimek-Chodacka等[44]通过构建双靶点CRISPR/Cas9载体分析了定向敲除黄烷酮-3-羟化酶基因(F3H)对胡萝卜Daucus carota紫色愈伤模型花青素积累的影响,同时比较了3种密码子优化的Cas9基因的编辑效率,发现3种Cas9基因均能够实现对靶基因的定向敲除,其中AteCas9最为有效。这些研究表明,CRISPR/Cas9技术具有广泛适用性,在园艺作物的基因功能研究和新品种选育中具有很强的应用价值和潜力。
2.3、 基因编辑技术在药用植物中的应用
药用植物种类很多,根据第三次全国重要资源普查结果,我国药用植物有11 146种[45],仅有200多种实现了人工栽培。受限于基因组信息的匮乏,目前在药用植物中应用基因编辑技术的报道较少。2016年,Kui等[46]选择木质素生物合成途径中的5个基因(C3H、C4H、4CL、CCR和IRX)作为靶基因,通过CRISPR/Cas9基因编辑技术对铁皮石斛Dendrobium officinale这5个靶基因实现定向突变,并进一步采用PCR扩增和测序技术验证突变,发现在靶基因的不同位点分别发生了核苷酸的取代、缺失和插入,证明了CRISPR/Cas9基因编辑技术能够在铁皮石斛中有效应用。可以进一步利用该技术对铁皮石斛其他途径进行修饰,并对基因功能和分子遗传进一步研究,有利于加快铁皮石斛分子育种进程。
Li等[47]采用CRISPR/Cas9基因编辑技术精确敲除丹参Salvia miltiorrhiza中参与生物合成的二萜合酶基因(SmCPS1),以发根农杆菌介导进行遗传转化获得了3个纯合和8个嵌合突变体。其中纯合丹参突变体中化学成分丹参酮,尤其是隐丹参酮、丹参酮IIA和丹参酮I完全缺失,而其他酚酸代谢物的合成不受影响;嵌合突变体中的丹参酮虽有减少但仍可检测到。Zhou等[48]利用CRISPR/Cas9系统对丹参中水溶性酚酸生物合成途径中的迷迭香酸合成酶(RAS)基因进行编辑,其获得的转基因丹参毛状根中RAS基因表达量降低进一步导致迷迭香酸(RA)、紫草酸B(LAB)含量显着降低,RA前体物3,4-二羟基苯乳酸(DHPL)含量显着升高。这些结果表明CRISPR/Cas9系统作为一种具有巨大应用潜力的工具在药用植物基因组编辑中的可行性,对进一步阐明丹参次生代谢产物合成途径中的关键酶基因功能,提高丹参的产量和质量具有重要意义。
苄基异喹啉生物碱(BIA)为罂粟Papaver somniferum中的特征性药效成分,在医学临床应用中,具有丰富的抗炎、抗菌、抗氧化、抗血小板凝集、抗心律失常、抗高血压、抗肿瘤等药理活性作用,如罂粟碱的解痉作用,吗啡的阵痛作用、小檗碱的降血脂作用等[49]。Alagoz等[50]使用CRISPR/Cas9系统通过非同源末端连接基因组修复敲除罂粟中调节BIAs生物合成的3'-羟基-N-甲基丙氨酸4'-O-甲基转移酶(4'OMT)基因,其敲除突变体中BIA的合成显着降低。由此可见,CRISPR/Cas9技术不仅可以研究药用植物的基因功能,还是药用植物代谢工程有效的研究工具。
3 、基因编辑在药用植物中的应用前景展望
当前,植物药在保护公众健康方面发挥着越来越重要的作用,尤其是在治疗一些疑难病、慢性病等方面,与化学药相比优势更为明显。然而,中药材生产中面临着各种问题,如部分名贵的野生药用植物濒临灭绝,栽培的药用植物产量和品质不稳定、抗病虫性降低,有些药效成分在药用植物中的含量很低,绝大多数药用植物对除草剂敏感,杂草防除困难等。高效的基因编辑修饰技术将在中药材产量、品质和抗性机制研究和中药材生产质量控制过程中发挥重要的作用。近年来,随着高通量测序技术的发展,越来越多的药用植物基因组序列信息获得了解析,功能基因组学和植物分子生物技术的发展为基因编辑技术在药用植物中的应用提供了充分的技术支持。基因编辑技术有望在以下几个方面促进药用植物的基础研究和生产应用。
3.1、 改善中药材的品质
中药材的品质包括外在品质、内在品质和加工品质等。品质性状的遗传受基因控制,并受环境条件影响;而药用植物药用部位发挥药效的物质基础为中药材含有的化学成分,化学成分的种类和含量决定了中药材的品质。中药材的化学成分复杂多样,主要有糖类、苷类、醌类化合物、苯丙素类化合物、黄酮类化合物、萜类和挥发油、生物碱、甾体类化合物、氨基酸等。中药材含有丰富的药效物质,同时,部分药材还含有一定的毒性成分,如生物碱类的麻黄碱、乌头碱、阿托品等;苷类成分强心苷、皂苷类、苦杏仁苷等[51]。改善药用植物的品质,一方面可通过基因编辑敲除抑制药效成分合成的基因来提高药效成分的含量,从而降低药用植物中有效成分的提取成本;另一方面则通过敲除合成毒性成分的基因降低有毒成分的含量,从而降低药材的不良反应。由于基因编辑技术仅修饰特定的基因,并不会导入外源基因片段,环境安全风险低,具备改善中药材品质的生产应用价值。
3.2 、解析药用植物药效成分合成的代谢通路
药用植物中的活性成分主要由植物次生代谢途径合成,其中经乙酸-丙二酸途径(AA-MA途径)生成脂肪酸类、酚类、醌类等化合物;萜类、甾体类化合物经甲戊二羟酸途径(MVA途径)合成;经莽草酸途径合成具有C6-C3和C6-C1基本结构的苯丙素类化合物;大多数的生物碱是由氨基酸途径合成;还可以通过以上复合途径合成次生代谢产物。通过代谢组学中的核磁共振(NMR)、质谱(MS)、色谱(HPLC、GC)及色谱质谱联用等技术手段对药用植物代谢物进行定性和定量分析,进一步结合基因组学和蛋白质组学阐明药用植物的次生代谢合成网络途径及关键酶的调控机制,明确药用植物次生代谢产物的合成积累规律和关键环节。药用植物药用部位药效成分的合成主要受其合成通路中的催化酶基因和转录因子调控,可利用基因编辑技术实现催化酶基因和转录因子的过量表达或敲除,通过检测代谢产物和目标成分的含量,进而明确药效成分合成的分子调控机制。
3.3、 提高药用植物产量
药用植物药用部位的产量组成包括生物产量和经济产量,经济产量主要是药用植物光合作用所形成的全部干物质产量;经济产量是指药用部位的产量。利用基因编辑技术提高药用植物产量可以从2个方面展开,其一,光合作用过程中将光能转化为有机物中稳定的化学能所涉及的两个反应阶段均需要一定酶的参与,通过基因编辑技术对光合作用过程中催化酶基因进行编辑,使相应的酶基因敲除或过表达从而对基因的功能进行研究,进一步调控基因的表达,从而提高光合作用产物。其二,利用基因编辑技术对控制不同药用部位的基因进行编辑,进一步获得药用部位产量高的药用植物。因此,在锁定药用植物药用部位产量控制关键功能基因的情况下,可利用基因编辑技术调控该基因的表达实现药用部位增产的目的。
3.4 、提高药用植物抗病虫性
病虫害是影响药用植物安全、优质生产的主要因素,选育抗病虫药用植物品种,提高药用植物抗病虫特性是防治病虫害,促进药用植物可持续发展的最经济、有效、安全的方法。根据其他植物领域中通过基因编辑获得抗病虫品种的研究成果,可以设计靶向敲除药用植物中病虫害易感性基因的CRISPR/Cas9基因编辑系统,或参考其他植物中已研究出的相关抗病虫基因并在药用植物基因组中比对获得抗性同源基因,进一步对该同源基因进行编辑,也可以尝试将其他植物中已报道的抗病虫基因,通过构建CRISPR表达载体转入药用植物中,从而提高药用植物的抗病虫性。
3.5 、提高药用植物除草剂抗性
在药用植物农业生产中普遍存在杂草危害严重的现象,人工除草成本很高。利用除草剂对不同植物类型的选择性防除,很多大田作物都实现了专用型除草剂的生产和应用。而大多数药用植物对除草剂非常敏感,喷施除草剂会对药用植物产生严重的药害作用,导致药用植物种子的发芽率降低、叶片卷曲和发黄,甚至导致药用植物萎蔫死亡,致使中药材农业生产上谈“除草剂”而色变。最新的研究表明,植物可通过自身基因的修饰获得对特定种类除草剂的抗性,如乙酰乳酸合酶基因ALS(对支链氨基酸的合成具有重要作用),对其单个氨基酸进行定向突变以降低植物对磺酰脲类除草剂的敏感性[52];5'-烯醇丙酮酰莽草酸-3-磷酸合酶基因EPSPS的突变体可以获得草甘膦抗性[53]。另外,通过基因编辑技术将除草剂解毒酶导入植物中,以降低除草剂对植物的伤害,从而获得除草剂抗性[54]。此外,相关研究还有报道出抗除草剂的其他潜在基因,如原卟啉原氧化酶(PPO)、α-微管蛋白等[55],可以对其进行进一步的研究明确其作用,为提高药用植物对除草剂的抗性奠定基础。
3.6、 加速药用植物的驯化
药用植物有10 000多种,然而,到目前为止绝大多数药用植物均以野生资源为主。野生药用植物存在分布分散、生境破坏严重、产量稳定性差等缺点,制约了植物药的持续供应和可持续发展。加速药用植物的驯化,有利于变野生药用植物为家种,保护濒危药用植物,保证中药材的永续利用。此外,加速药用植物野生种质资源的驯化,能够进一步对药用植物进行统一化栽培管理,对药用植物的不良性状进行驯化。利用传统的栽培和育种技术驯化野生植物,周期十分漫长,获得理想的栽培性状常常需要数年甚至数十年。而近年来发展起来的基因编辑技术为快速驯化野生药用植物提供了可能。Li等[56]通过基因编辑技术靶向番茄分枝数量基因、果实大小基因等,获得的突变体植株在保留野生番茄优良性状的前提下改善了番茄的不良性状。因此,可利用基因编辑技术对药用植物中调控不良性状的基因进行修饰,定向驯化农艺性状,从而实现药用植物的快速驯化,加速中药材的现代化发展进程。
参考文献
[1] ZHANG Y, MASSEL K, GODWIN I D, et al. Applications and potential of genome editing in crop improvement[J]. Genome Biol, 2018, 19(1): 210.
[2] 肖安, 胡莹莹, 王唯晔, 等. 人工锌指核酸酶介导的基因组定点修饰技术[J]. 遗传, 2011, 33(7): 3-21.
[3] 沈延, 肖安, 黄鹏, 等. 类转录激活因子效应物核酸酶(TALEN)介导的基因组定点修饰技术[J]. 遗传, 2013, 35(4): 295-309.
[4] SOREK R, KUNIN V, HUGENHOLTZ P. CRISPR — a widespread system that provides acquired resistance against phages in bacteria and archaea[J]. Nat Rev Microbiol, 2008, 6(3): 181.
[5] DOUDNA J A, CHARPENTIER E. The new frontier of genome engineering with CRISPR-Cas9[J]. Science, 2014, 346(6213): 1258096.
[6] CHUGUNOVA A A, Dontsova O A, Sergiev P V. Methods of genome engineering: a new era of molecular biology[J]. Biochemistry (Moscow), 2016, 81(7): 662-677.
[7] BIBIKOVA M, BEUMER K, TRAUTMAN J K, et al. Enhancing gene targeting with designed zinc finger nucleases[J]. Science, 2003, 300(5620): 764-764.
[8] VANAMEE ? S, SANTAGATA S, AGGARWAL A K. FokI requires two specific DNA sites for cleavage[J]. J Mol Biol, 2001, 309(1): 69-78.
[9] DURAI S, MANI M, KANDAVELOU K, et al. Zinc finger nucleases: custom-designed molecular scissors for genome engineering of plant and mammalian cells[J]. Nucleic Acids Res, 2005, 33(18): 5978-5990.
[10] MOSCOU M J, BOGDANOVE A J. A simple cipher governs DNA recognition by TAL effectors[J]. Science, 2009, 326(5959): 1501-1501.
[11] STREUBEL J, BL?CHER C, LANDGRAF A, et al. TAL effector RVD specificities and efficiencies[J]. Nat Biotechnol, 2012, 30(7): 593.
[12] 李君, 张毅, 陈坤玲, 等. CRISPR/Cas系统: RNA靶向的基因组定向编辑新技术[J]. 遗传, 2013, 35(11): 1265-1273.
[13] BHAYA D, DAVISON M, BARRANGOU R. CRISPR-Cas systems in bacteria and archaea: versatile small RNAs for adaptive defense and regulation[J]. Annu Rev Genet, 2011, 45: 273-297.
[14] JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821.
[15] JANSEN R, EMBDEN J D A, GAASTRA W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-1575.
[16] KARGINOV F V, HANNON G J. The CRISPR system: small RNA-guided defense in bacteria and archaea[J]. Molecular cell, 2010, 37(1): 7-19.
[17] WIEDENHEFT B, STERNBERG S H, DOUDNA J A. RNA-guided genetic silencing systems in bacteria and archaea[J]. Nature, 2012, 482(7385): 331.
[18] RAN F A, HSU P D, LIN C Y, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1380-1389.
[19] 钟成, 黄龙辉, 刘其敬, 等. CRISPR/dCas9在细菌转录调控中的应用及展望[J]. 天津科技大学学报, 2019, 34(2): 1-5+11.
[20] ZETSCHE B, GOOTENBERG J S, ABUDAYYEH O O, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell, 2015, 163(3): 759-771.
[21] ZETSCHE B, HEIDENREICH M, MOHANRAJU P, et al. Multiplex gene editing by CRISPR-Cpf1 using a single crRNA array[J]. Nat Biotechnol, 2017, 35(1): 31.
[22] LI T, LIU B, SPALDING M H, et al. High-efficiency TALEN-based gene editing produces disease-resistant rice[J]. Nature biotechnology, 2012, 30(5): 390.
[23] 姜明君, 常振仪, 卢嘉威, 等. 应用TALEN技术定点突变水稻苯达松抗性基因CYP81A6[J]. 农业生物技术学报, 2016, 24(8): 1225-1232.
[24] SUN Y, ZHANG X, WU C, et al. Engineering herbicide-resistant rice plants through CRISPR/Cas9-mediated homologous recombination of acetolactate synthase[J]. Mol Plant, 2016, 9(4): 628-631.
[25] WANG F, WANG C, LIU P, et al. Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor gene OsERF922[J]. PloS one, 2016, 11(4): e0154027.
[26] LI J, MENG X, ZONG Y, et al. Gene replacements and insertions in rice by intron targeting using CRISPR–Cas9[J]. Nat Plants, 2016, 2(10): 16139.
[27] 邵高能, 谢黎虹, 焦桂爱, 等. 利用CRISPR/CAS9技术编辑水稻香味基因Badh2[J]. 中国水稻科学, 2017, 31(2): 216-222.
[28] 王加峰, 郑才敏, 刘维, 等. 基于CRISPR/Cas9技术的水稻千粒重基因tgw6突变体的创建[J]. 作物学报, 2016, 42(8): 1160-1167.
[29] SHUKLA V K, DOYON Y, MILLER J C, et al. Precise genome modification in the crop species Zea mays using zinc-finger nucleases[J]. Nature, 2009, 459(7245): 437.
[30] CHAR S N, UNGER-WALLACE E, FRAME B, et al. Heritable site-specific mutagenesis using TALENs in maize[J]. Plant Biotechnol J, 2015, 13(7): 1002-1010.
[31] SHI J, GAO H, WANG H, et al. ARGOS 8 variants generated by CRISPR-Cas9 improve maize grain yield under field drought stress conditions[J]. Plant Biotechnol J, 2017, 15(2): 207-216.
[32] DU H, ZENG X, ZHAO M, et al. Efficient targeted mutagenesis in soybean by TALENs and CRISPR/Cas9[J]. J Biotechnol, 2016, 217: 90-97.
[33] KIM D, ALPTEKIN B, BUDAK H. CRISPR/Cas9 genome editing in wheat[J]. Funct Integr Genomic, 2018, 18(1): 31-41.
[34] WENDT T, HOLM P B, STARKER C G, et al. TAL effector nucleases induce mutations at a pre-selected location in the genome of primary barley transformants[J]. Plant Mol Biol, 2013, 83(3): 279-285.
[35] GAO W, LONG L, TIAN X, et al. Genome editing in cotton with the CRISPR/Cas9 system[J]. Front Plant Sci, 2017, 8: 1364.
[36] LOR V S, STARKER C G, VOYTAS D F, et al. Targeted mutagenesis of the tomato PROCERA gene using transcription activator-like effector nucleases[J]. Plant Physiol, 2014, 166(3): 1288-1291.
[37] ?ERM?K T, BALTES N J, ?EGAN R, et al. High-frequency, precise modification of the tomato genome[J]. Genome Biol, 2015, 16(1): 232.
[38] ITO Y, NISHIZAWA-YOKOI A, ENDO M, et al. CRISPR/Cas9-mediated mutagenesis of the RIN locus that regulates tomato fruit ripening[J]. Biochem Bioph Res Co, 2015, 467(1): 76-82.
[39] UETA R, ABE C, WATANABE T, et al. Rapid breeding of parthenocarpic tomato plants using CRISPR/Cas9[J]. Scientific reports, 2017, 7(1): 507.
[40] YANG T X, Deng L, Zhao W, et al. Rapid breeding of pink-fruited tomato hybrids using CRISPR/Cas9 system[J]. J Genet Genomics, 2019, 46(10): 505-508.
[41] FORSYTH A, WEEKS T, RICHAEL C, et al. Transcription activator-like effector nucleases (TALEN)-mediated targeted DNA insertion in potato plants[J]. Front Plant Sci, 2016, 7: 1572.
[42] KANNAN B, JUNG J H, MOXLEY G W, et al. TALEN-mediated targeted mutagenesis of more than 100 COMT copies/alleles in highly polyploid sugarcane improves saccharification efficiency without compromising biomass yield[J]. Plant Biotechnol J, 2018, 16(4): 856-866.
[43] TIAN S, JIANG L, GAO Q, et al. Efficient CRISPR/Cas9-based gene knockout in watermelon[J]. Plant Cell Rep, 2017, 36(3): 399-406.
[44] KLIMEK-CHODACKA M , Oleszkiewicz T , Lowder L G , et al. Efficient CRISPR/Cas9-based genome editing in carrot cells[J]. Plant Cell Rep, 2018, 37(4):575–586.
[45] 余椿生. 全国中药资源普查通过验收[J]. 中药材, 1995, 18(5):265.
[46] KUI L,CHEN H,ZHANG W,et al. Building a genetic manipulation tool box for orchid biology: identification of constitutive promoters and application of CRISPR/Cas9 in the orchid, Dendrobium officinale[J]. Front Plant Sci, 2016, 7: 2036.
[47] LI B, CUI G, SHEN G, et al. Targeted mutagenesis in the medicinal plant Salvia miltiorrhiza[J]. Sci Rep, 2017, 7: 43320.
[48] ZHOU Z, TAN H, LI Q, et al. CRISPR/Cas9-mediated efficient targeted mutagenesis of RAS in Salvia miltiorrhiza[J]. Phytochemistry, 2018, 148: 63-70.
[49] 李丹, 蒋建勤. 苄基异喹啉类生物碱的药理活性研究进展[J]. 广东化工, 2017, 44(9): 141-142.
[50] ALAGOZ Y, GURKOK T, ZHANG B, et al. Manipulating the biosynthesis of bioactive compound alkaloids for next-generation metabolic engineering in opium poppy using CRISPR-Cas 9 genome editing technology[J]. Sci Rep, 2016, 6: 30910.
[51] 孙圆媛, 高月. 中药毒性的研究概况[J]. 航空航天医药, 2003, 14(4): 253-255.
[52] 王秀君, 郎志宏, 单安山,等. 氨基酸生物合成抑制剂类除草剂作用机理及耐除草剂转基因植物研究进展[J]. 中国生物工程杂志, 2008, 28(2): 110-116.
[53] 李燕敏, 祁显涛, 刘昌林, 等. 除草剂抗性农作物育种研究进展[J]. 作物杂志, 2017,(2): 1-6.
[54] 阮燕晔, 薛寥莎. 植物抗除草剂基因研究进展[J]. 南方农业学报, 2012, 43(4): 462-466.
[55] 张杰, 卢百亨. 非转基因抗除草剂作物的获得方法研究[J]. 乡村科技, 2018, (13): 89-91.
[56] LI T, YANG X, YU Y, et al. Domestication of wild tomato is accelerated by genome editing[J]. Nat Biotechnol, 2018, 36(12): 1160-1163.





