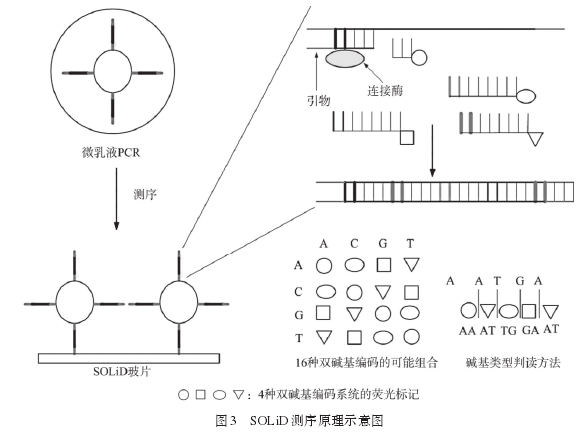
1.3 SOLi D测序平台
SOLi D测序技术由美国Agencourt公司开发,2006年被ABI公司收购。ABI于2007推出SOLi D的第一个测序平台,2010年又推出SOLi D 5500XL.同Roche 454测序平台一样,SOLi D也采用微乳液PCR,不同之处在于其采用寡核苷酸连接测序。SOLi D测序原理见图3.微乳液PCR后模板链经磁珠连接至SOLi D玻片上进行测序反应。首先,通用引物与模版链上的接头互补配对,加入16种8碱基探针和连接酶竞争与引物连接的位点。连接反应后,探针的后3个碱基被清除,荧光信号释放,多次加入底物直至延伸至待测链末端,然后换用新引物进行新一轮测序反应。新引物与第一个引物区别是长度相同、与接头配对位置相差一个碱基,完成待测链测序需要5个此种引物。采用双碱基编码策略进行连接反应,即每两个相邻位点的碱基对应16种任意组合的8碱基探针中的4种荧光信号之一,核苷酸链延伸时,每个位点被扫描2次,准确率达99.94%,但双碱基编码策略也可能导致连锁解码错误。SOLi D一次循环可产生75~110 bp读长,产生300 Gb的测序序列。同Illumina一样,SOLi D可用于全基因组重测序确定单核苷酸多态性、缺失和插入等基因组结构变异,也可用于目标区域捕获测序、染色质免疫共沉淀测序和RAN测序,但读长短、成本高和数据结果分析困难的不足使其应用受限。
1.4 Ion Torrent PGM和Proton半导体测序
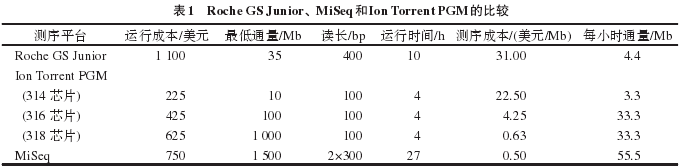
美国Ion Torrent公司自2010年被Life Technolo-gies收购后陆续推出Ion Torrent PGM(2010年) 和Proton(2012年),二者是介于第二代和第三代之间的测序平台,其核心技术是Ion Torrent公司开发的半导体测序[14].半导体测序技术也采用微乳液PCR,不同之处在于其检测的是单核苷酸与芯片上固定的模板链配对时释放氢离子引起的p H变化,而不是荧光信号。Ion Torrent PGM有3种芯片,314芯片适合小基因组测序,316和318芯片用于全转录组测序和染色质免疫共沉淀测序[15].Ion Torrent PGM通量虽低,但速度快,成本低且仪器规模小,因此应用广泛,适合16s RNA测序[16]、微生物和病毒的从头测序和重测序、目标区域捕获测序、单核苷酸多态性检测[17]、短串联重复序列测序[18]、混合感染鉴定和线粒体DNA测序等[19- 20].但Ion Torrent PGM也存在因多次洗脱过程导致的错误累积、阅读高度重复序列和同种多聚序列时出错率高等不足。现有Roche GS Junior、Ion Tor-rent PGM和Mi Seq 3种针对临床和中小型实验室的小型测序仪,其性能比较见表1[21].
2第三代测序技术
第三代测序技术是在第二代基础上增加读长,降低试剂成本,并且加快运行速度。其显着特点是单分子测序,即不经PCR直接进行边合成边测序[22],不仅简化了样品处理过程,避免了扩增可能引入的错配,而且不受鸟嘌呤和胞嘧啶或腺嘌呤和胸腺嘧啶含量的影响,因此第三代测序技术能直接对RNA和甲基化DNA序列进行测序[23].现有的第三代测序平台包括美国Helicos Bioscience公司的Heli Scope遗传分析系统和Pacific Biosciences公司的Pac Bio RS单分子实时测序系统。
2.1 Heli Scope遗传分析系统
Heli Scope遗传分析系统为第一个单分子测序系统,由美国Helicos Bioscience公司于2008年推出。其基本过程为3’末端多聚腺嘌呤修饰的单链DNA模板被芯片上多聚胸腺嘧啶修饰的引物捕获,在DNA聚合酶的作用下,荧光标记的d NTP与模板链配对,通过采集荧光信号可获得碱基信息[24].该系统每次循环可产生21~25 Gb,平均读长35 bp的序列。Heli Scope遗传分析系统所需样本量较少且对样本质量要求低,可用于古生物信息检测[25].Heli Scope遗传分析系统最常用于基因表达分析,如在哺乳动物基因功能注解计划中用Heli-Scope进行基因表达加帽分析[26-27].
2.2 Pac Bio RS单分子实时测序系统
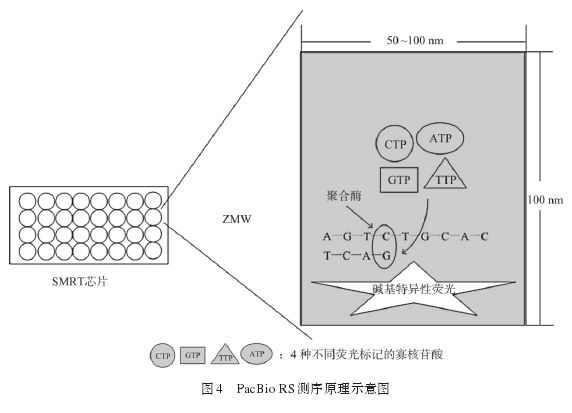
Pac Bio RS单分子实时测序系统由美国PacificBiosciences公司于2010年推出,采用四色荧光标记的d NTP和单分子实时芯片上的零级波导 (zero-mode waveguides,ZMW)对单个DNA分子进行测序。ZWM是一种直径50~100 nm、深度100 nm的孔状纳米光电结构,当光线进入后呈指数衰减,仅靠近基底的部分被照亮。DNA聚合酶固定在ZMW底部,加入模版、引物和四色荧光标记的d NTP后进行DNA合成,只有参加反应的d NTP才能停留在ZMW底部,从而使d NTP的荧光信号被识别[28],该测序过程见图4.Pac Bio RS单分子实时测序系统的优势在于读长,最新的Pac Bio RSⅡ读长可达20 kb.Pac Bio RS每次循环产生400 Mb序列,成本为2~17美元/ Mb,与其他NGS平台相比,其通量低,成本高,且单碱基识别错误率高达14%[7],但可通过提高循环次数来改善,也可与第二代测序技术联合应用以降低成本并提高准确度[29].Rhoads等[30]最近的研究表明Pac Bio RS适合于从头测序、基因结构突变检测、复杂重复序列测序、发现基因亚型、4-甲基胞嘧啶和6-甲基腺嘌呤检测。