摘 要: 本文以C57BL/6J小鼠为模式动物,研究高脂饮食对不同性别小鼠肠道微生物的影响。16只8周龄SPF级C57BL/6J小鼠,雌雄各半,以基础饲料喂养的小鼠为对照组,45%高脂饲料喂养的小鼠为实验组(每组4只)。饮食干预6周后,收集小鼠新鲜粪便并提取总基因组DNA,通过PCR扩增16S-rDNA的V3+V4区域并进行HiSeq2500高通量测序,分析菌群的组成及丰富度变化。结果显示,高脂饮食改变了小鼠肠道微生物组成及其多样性,在门分类水平上,较之对照组,雌性高脂组小鼠的变形菌门相对丰富度显着下降(P<0.05),放线菌门相对丰富度显着升高(P<0.001),雄性高脂组小鼠的变形菌门相对丰富度显着上升(P<0.001),放线菌门相对丰富度无明显变化。在种分类水平上,雌性高脂组小鼠的Lactobacillus_murinus菌种相对丰富度与对照组相比显着升高(P<0.01),雄性高脂组较之对照组无显着差异。表明高脂饮食对雌性和雄性小鼠肠道微生物组成及结构的影响不同。本研究结果为深入研究饮食及性别因素对肠道微生物的影响提供依据。
关键词: 高脂饮食; 性别; 肠道微生物; 微生物多样性;
Abstract: In this work, the impact of high fat diet on gut microbiota in the male and female C57BL/6J mice was investigated. Sixteen 8-week- old C57BL/6J mice (8 per sex) were housed in the specific pathogen free (SPF) condition. The mice fed with a normal diet were used as the control groups, fed with a 45% kcal fat diet were used as the experimental groups (4 mice in each group). After 6 weeks of dietary intervention, fresh fecal samples were collected from each mice. For high throughput HiSeq 2500 sequencing analysis, fecal bacterial genomic DNA was extracted and amplified based on the V3+V4 region of the bacterial 16S-rDNA gene by PCR (Polymerase Chain Reaction) The changes in composition and relative abundance of gut microbiota were evaluated. The results showed that high-fat diet altered gut microbiota composition affecting the diversity and similarity. In the female high-fat diet group, the relative abundance of Proteobacteria phylum significantly decreased (P<0.05) and the Actinobacteria phylum significantly increased (P<0.001) compared with the control diet group. However, in the male high-fat diet group, the relative abundance of Proteobacteria phylum significantly increased (P<0.001) and the Actinobacteria phylum was no obvious change compared with the control diet group. Moreover, the relative abundance of L.murinus was significantly higher in the high-fat diet group than that in the control diet group of female mice (P<0.01); however, this species of bacteria did not show significant change in the male high-fat diet group. The effects of high-fat diet on the composition and structure of gut microbiota were different in the female and male mice, these results provide data for further studying the effects of dietary and gender factors on intestinal microorganisms.
Keyword: High-fat diet; Gender; Gut microbiota; Microbiota diversity;
肠道微生物作为“人类第二基因组”[1],数量庞大、种类繁多,不同种类的肠道微生物相互作用,共同调控宿主的免疫功能,维持宿主肠道内环境的稳定[2]。人类和小鼠肠道微生物组成相似,主要分为厚壁菌门、拟杆菌门、放线菌门及变形菌门等[3]。正常情况下,肠道微生物与宿主及外部环境处于平衡状态,并在能量调节、营养吸收和免疫调节等方面发挥着重要的作用[4],一旦平衡遭到破坏,引起肠道菌群失调,将导致宿主代谢紊乱,对宿主健康造成威胁。因此,了解肠道微生物的组成结构及其变化,对于维护宿主健康及疾病预防诊断至关重要。
众所周知,膳食结构[5]、遗传因素[6]、分娩方式[7]、哺乳方式[8,9]、抗生素和药物的使用[10,11]等都会影响肠道微生物的组成,其中膳食结构发生改变的24小时内,肠道微生物的组成就会发生显着变化[12]。研究人员对89种近交系小鼠的肠道微生物进行了独立分析,发现各品系小鼠的肠道微生物组成和多样性均有明显的差异,虽然性别对肠道微生物的影响不及宿主基因型[13],但提供了性别影响肠道微生物组成差异的暗示性证据。另外,一项对75名男性和绝经后女性的肠道菌群的研究表明,在不考虑身体质量指数的情况下,微生物群落多样性和总体组成没有显着性差异[14]。然而另一项对1135名男性和女性的研究中,不同性别人群肠道微生物的组成有显着差异[15]。总之,对动物和人类的研究均表明,性别也是影响肠道微生物的重要因素[13,16]。
近年来,随着生活水平的不断提高,人类的饮食习惯越来越趋向于“西方化”的高脂、高糖膳食。据文献报道,饮食结构与宿主性别共同作用,影响肠道微生物的组成[17]。而高脂饮食诱导雌性和雄性宿主肠道微生物的变化情况或趋势是否一致尚不明确[18]。因此,为了研究高脂饮食对雌性和雄性小鼠肠道微生物组成结构的影响,以不同性别的C57BL/6J小鼠为研究对象,分别用基础饲料和高脂饲料饲喂,饮食干预6周后收集小鼠的新鲜粪便,对小鼠粪便细菌16S-rDNA的V3+V4区域进行测序,分析比较雌性和雄性小鼠肠道微生物菌群的组成结构,探讨高脂饮食对不同性别小鼠肠道微生物的影响,为深入研究饮食及性别因素对肠道微生物的影响提供依据。
1、 材料与方法
1.1 、材料与仪器
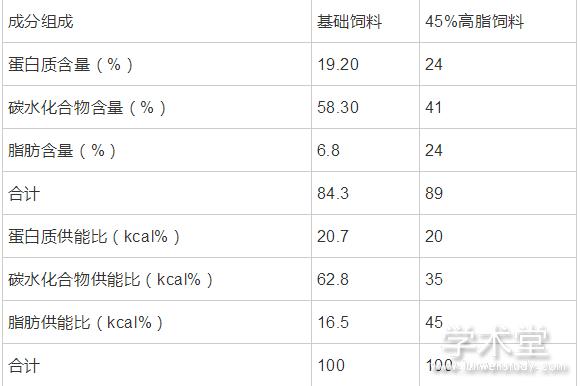
实验动物 16只6周龄SPF级C57BL/6J小鼠(动物生产许可证号:SCXK(苏)2018-0008),雌雄各半,体重15 ~ 18 g,购自南京大学动物模式研究所。基础饲料(舒克贝塔1010013) 江苏省协同医药生物工程有限责任公司;45 %高脂饲料(Research Diets, D12451) 北京凯国科技有限公司;饲料主要物质供能比见表1。E.Z.N.A Stool DNA Kit试剂盒 美国Omega Bio-Tek公司;琼脂糖粉末 北京沃比森科技公司;AxyPrep DNA Gel Extraction Kit 美国Axygen Biosciences公司;引物341F/806R由生工生物公司合成;KOD Buffer/ dNTPs /KOD聚合酶 宝日医生物技术(北京)有限公司。
表1 饲料主要物质供能表
微量移液器 德国Eppendorf公司;普通PCR仪 美国Bio-rad公司;电泳仪 美国Bio-rad公司;液氮罐 美国Thermo公司;凝胶成像仪 美国Bio-rad公司;QuantiFluor-ST荧光计 美国Promega公司;低温台式高性能离心机 美国Thermo公司;Milli-Q纯水仪 美国Millipore公司;超低温冰箱 长虹美菱股份有限公司;全自动雪花制冰机 南京先欧科技有限公司。
1.2、 实验方法
1.2.1、 动物模型制备
C57BL/6J小鼠按雌雄各随机分为对照组(F-CON&M-CON)和高脂组(F-HF&M-HF),每组4只小鼠,基础饲料适应性喂养2周后,分别饲喂基础饲料(CON)和45%高脂饲料(HF),饲养环境为昼夜12 h交替,控制室内温度在22~25 ℃,湿度在55% ~ 65%,食物和水自由摄取。
1.2.2 、样本采集
饮食干预六周后,收集每组小鼠新鲜清洁无污染的粪便样本,迅速置于液氮中,并于-80℃保存备用。
1.2.3 、肠道微生物DNA提取及PCR扩增
按照E.Z.N.A Stool DNA Kit试剂盒(Omega Biotek,Norcross,GA,U.S.)提取粪便DNA样品,对提取后的DNA产物经琼脂糖凝胶电泳进行检测。然后用带有barcode的特异引物扩增16S-rDNA的V3+V4区域。引物序列为341F:CCTACGGGNGGCWGCAG;806R:GGACTACHVGGGTATCTAAT。扩增体系为:5 μL的10 × KOD Buffer,5 μL的2.5 mM dNTPs,1.5 μL引物(5 μM), 1 μL的KOD聚合酶和100 ng模板DNA,最终体积为50 μL。扩增条件如下:95℃预变性2min,随后 98℃ 变性10 s,62℃ 退火30 s,68℃延伸30 s,共27个循环,最后68℃延伸10min。
1.2.4 、建库和测序
从2%琼脂糖凝胶中回收扩增产物,用AxyPrep DNA Gel Extraction Kit (Axygen Biosciences, Union City, GA, U.S.)纯化扩增产物,再经QuantiFluor-ST (Promega,U.S.)荧光计进行定量。将纯化的扩增产物等量混合,连接测序接头,根据Illumina说明构建测序文库,以Hiseq2500的PE250模式上机测序,将得到的原始Reads存储到NCBI序列读存档(SRA,Sequence Read Archive)数据库中用于后续分析[19]。
1.2.5、 数据处理
利用Mothur (v1.39.1) 软件对tag序列进行去冗余处理[20],从中挑选出 unique tag 序列,然后用 Uparse (v9.2.64_i86linux32) 软件对所有样品的全部 Effective Tags 序列按97%的相似性聚类成为OTUs (Operational Taxonomic Units) ,再与RDP (Ribosomal Database Project) 数据库进行比对,对OTUs进行物种注释[21]。基于OTU的丰度结果,利用R语言中的GUniFrac (v1.0) 计算两两样本间的Weighted Unifrac和Unweighted Unifrac距离[22],然后进行多元统计的主坐标分析(PCoA ,Principal coordinates analysis)和非加权组平均聚类分析(UPGMA,Unweighted Pair-group Method with Arithmetic Means)。
1.3、 统计学分析
利用GraphPad Prism7.0统计学软件对图像分析的数据进行统计学分析,所有数据的分析结果均以平均数±标准误差(Mean ± SED)表示,两组间的比较采用t检验(t-test),P<0.05为差异显着,有统计学意义。
2、 结果与分析
2.1、 高脂饮食对小鼠肠道微生物组成的影响
由图1A可知,饮食干预6周后,小鼠体重已有明显差异,同一性别小鼠,高脂饮食组体重显着高于基础饮食组体重(P<0.05)。OTUs主坐标分析结果表明,基础饮食小鼠和高脂饮食小鼠肠道微生物的数据点表现出各自聚集分布的情况,并且膳食因素对肠道微生物差异的贡献值达52.46%(图1B)。说明饮食干预影响了小鼠肠道微生物组成,这可能与营养物质的吸收以及能量代谢水平差异有关。
图1不同饮食组小鼠体重和肠道微生物主坐标分析
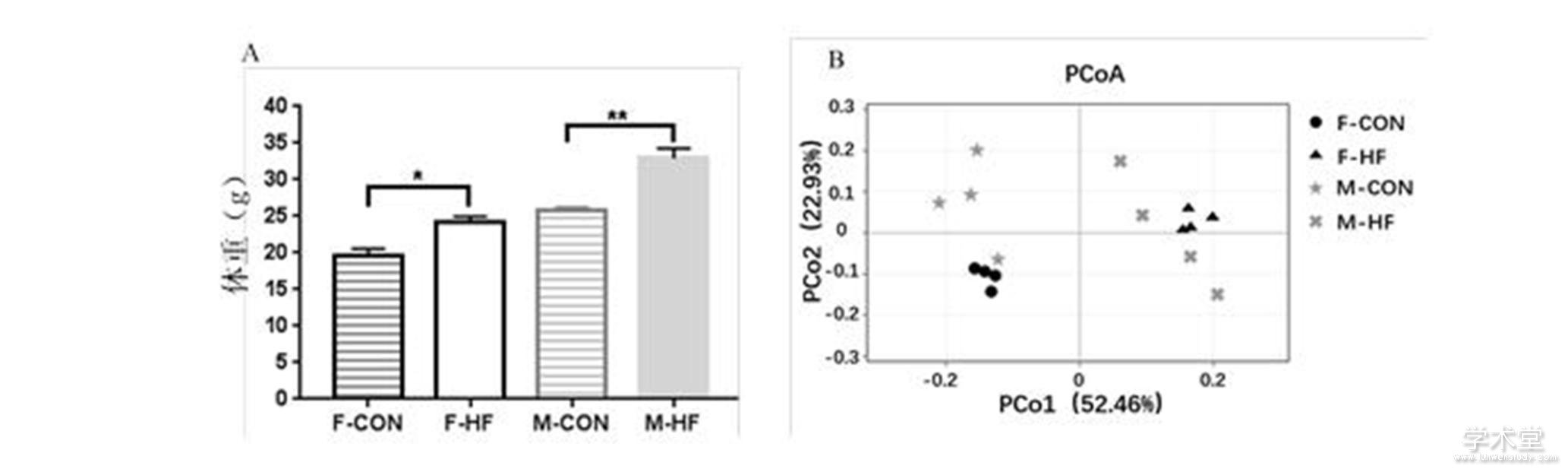
Fig.1 The body weight and the principal coordinates analysis(PCoA)of gut microbiota in different diet groups
注:F-CON代表雌性基础饲料组,F-HF代表雌性高脂组;M-CON代表雄性基础饲料组,M-HF代表雄性高脂组;不同形状表示不同的饮食组;n=4,与对照组相比,* P<0.05, ** P<0.01
2.2、 高脂饮食对小鼠肠道微生物多样性的影响
由图2可知,对照组小鼠和高脂组小鼠肠道微生物以组为单位聚集在同一簇;在对照组中,雌性小鼠和雄性小鼠肠道微生物各自成簇,高脂组小鼠肠道微生物也基于性别各自成簇,表明性别也是影响肠道微生物多样性的因素。相同饮食的小鼠之间的共同分支距离更短且空间距离更近,意味着肠道微生物更相似,说明高脂饮食改变了雌性和雄性小鼠肠道微生物多样性,同时也进一步证实了饮食结构和性别是影响肠道微生物多样性的重要因素。
图2 不同饮食组小鼠的肠道微生物UPGMA分类树

Fig.2 The UPGMA cluster dendrogram of gut microbiota in different diet groups
注:每个分支对应一个样本,分支距离代表相似性,距离越短,样品越相似
2.3、高脂饮食对雌性和雄性小鼠肠道微生物菌门组成的影响
在门分类水平上,对小鼠肠道微生物组成的分析发现,各组小鼠优势菌均由厚壁菌门、拟杆菌门、变形菌门和放线菌门组成。如表2所示,高脂饮食条件下,雌性小鼠变形菌门相对丰富度为(5.46 ± 0.55)%,显着低于对照组(P<0.05);而雄性小鼠变形菌门的相对丰富度为(7.86 ± 0.68)%,相比于对照组(3.01± 0.31)%极显着升高(P<0.001)。另外,高脂饮食条件下,雌性小鼠放线菌门的相对丰富度(8.57 ± 1.15)%与对照组(0.78 ± 0.067)%相比极显着升高(P<0.001),而雄性小鼠放线菌门相对丰富度(2.92 ± 0.66)%与对照组(3.99 ± 2.30)%相比无显着下降(P>0.05)。以上数据表明高脂饮食对不同性别小鼠肠道菌群产生不同的影响,推断不同菌门在宿主体内可能有不同的代谢偏好,并且变形菌门对饮食干预的反映较为显着,这也许是由于菌群与性激素水平之间的相互作用,继而调节肠道微生态,但具体机制还需进一步研究。
表2 门分类水平优势菌门的相对丰富度(%)
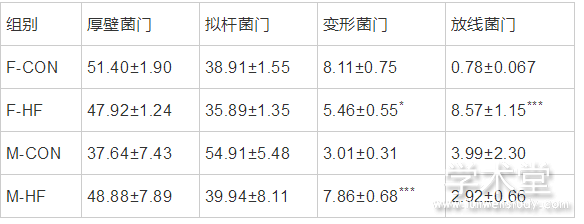
注:与相同性别对照组相比,* P< 0.05; *** P<0.001
2.4、高脂饮食对雌性和雄性小鼠肠道微生物菌种组成的影响
由图3A可知,在种分类水平上各组小鼠肠道微生物都能够注释到的物种主要包括Lactobacillus_murinus、Lachnospiraceae_bacterium_615、Blautia_coccoides、Leptum、Bacteroides_acidifaciens等菌种。如图3B所示,高脂饮食干预下,厚壁菌门分类下的L. murinus菌种在雌性小鼠的相对丰富度为(3.13 ± 0.28)%,与对照组(1.55 ± 0.23)%相比显着上升(P<0.01),但该菌种在雄性小鼠中呈相反趋势,高脂饮食下其相对丰富度为(1.27 ± 0.22)%,与对照组(3.88± 1.44)%相比虽无显着性差异,但呈下降趋势,进一步证实高脂饮食干预下,肠道微生物因宿主性别不同而发生不同的变化。
图3 小鼠肠道微生物种水平分布及差异情况
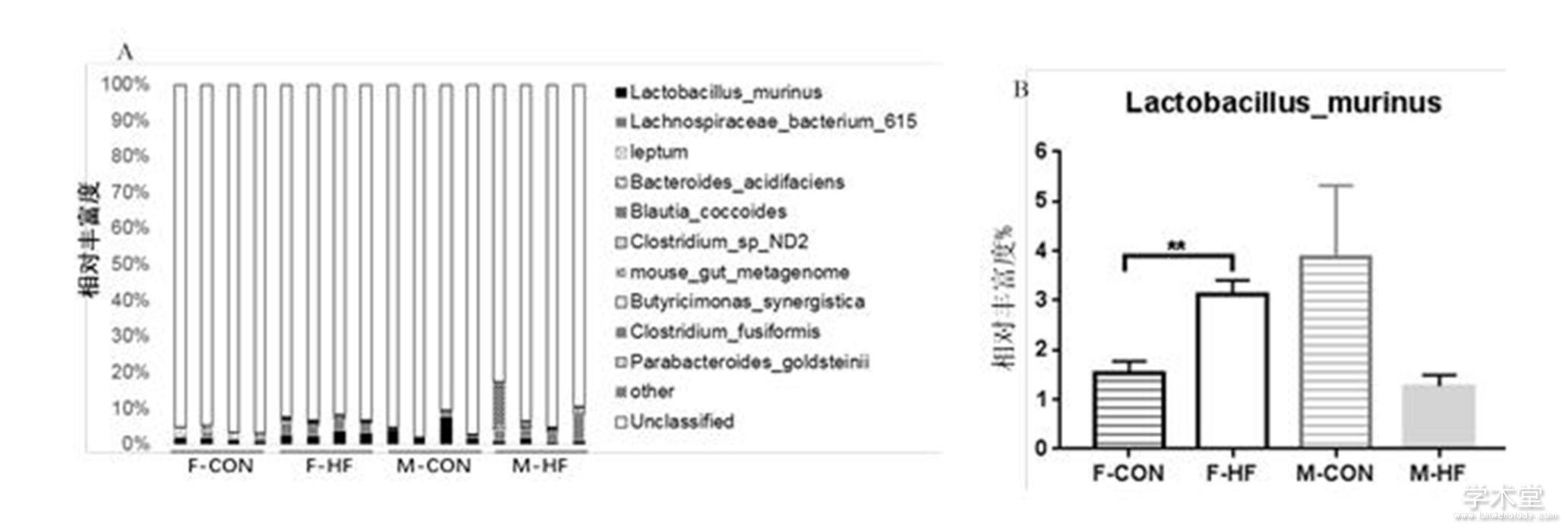
Fig.3 The composition and relative abundance of gut microbiota at species level of mice
注:与对照组相比,** P<0.01
3、 讨论
本研究使用45% 高脂饲料饲喂8周龄雌性和雄性小鼠,模拟当前人类“西方化”的高脂饮食习惯。据文献报道,成人体内重要的肠道微生物包括厚壁菌门、拟杆菌门、放线菌门和变形菌门等[3],本研究结果显示,在门分类水平上,所有小鼠的优势菌门也是由这4大类组成。目前关于性别对肠道菌群组成的影响的报道,结果一直不一致[13,17,23,24],考虑是由于饮食、宿主遗传背景和年龄等因素在一定程度上的影响所致。本研究旨在探讨高脂饮食与性别对肠道微生物的不同影响。显然,在遗传背景和年龄相同的情况下,饮食和性别是影响肠道微生物组成的重要因素。Carmody R等[25]对五种近交系小鼠的研究发现:小鼠的肠道微生因饮食波动呈线性剂量关系,表明饮食在宿主肠道微生物差异中起到了主导作用,本研究也充分表明了高脂饮食对肠道微生物菌群的影响显着(贡献值达52.46%)。此外,在高脂饮食条件下,雄性小鼠和雄性小鼠变形菌门出现相反的变化,此种差异可能是由于宿主体内激素水平差异引起菌门代谢偏好所致[26]。另有研究表明,肥胖女性体内的放线菌门丰富度升高[27],本研究也发现:在高脂饮食条件下,雌性小鼠放线菌门的丰富度显着升高,但雄性小鼠却无显着变化,这可能与宿主能量吸收及代谢水平有关[28]。进一步分析种分类水平上的菌种差异,发现在高脂饮食条件下,厚壁菌门下的Lactobacillus_ murinus菌种在雌性小鼠中显着上升,但在雄性小鼠中无显着变化,Pan等[29]发现L. murinus菌种能够降低无菌小鼠的肠道通透性和血清内毒素水平,从而减轻衰老菌群引起的炎症反应,说明该菌种具有一定的抗炎作用;另有研究表明L.murinus菌种并不会诱导C57BL/6小鼠肥胖[30],因此该类菌种与肥胖产生的联系需要进一步验证。
4、 结论
本研究证实了高脂饮食可以改变雌性和雄性小鼠肠道微生态的结构,但对雌性和雄性小鼠肠道微生物组成及变化的影响并不完全相同,为深入研究饮食及性别因素对肠道微生物的影响提供依据。然而,研究结果并不能解释这种差异是否与激素水平、能量代谢等因素相关,因此还有待进一步研究。
参考文献
[1] Zmora N, Zeevi D, Korem T, et al. Taking it personally: personalized utilization of the human microbiome in health and disease[J]. Cell Host Microbe. 2016, 19(1): 12-20.
[2] Rosenbaum M, Knight R, Leibel R L. The gut microbiota in human energy homeostasis and obesity[J]. Trends in Endocrinology & Metabolism. 2015, 26(9): 493-501.
[3] Hillman E T, Lu H, Yao T, et al. Microbial ecology along the gastrointestinal tract[J]. Microbes and environments. 2017, 32(4): 300-313.
[4] Maruvada P, Leone V, Kaplan L M, et al. The Human Microbiome and Obesity: Moving beyond Associations.[J]. Cell Host & Microbe. 2017, 22(5): 589-599.
[5] Turnbaugh P J, Ridaura V K, Faith J J, et al. The effect of diet on the human gut microbiome: A metagenomic analysis in humanized gnotobiotic mice[J]. Science Translational Medicine. 2009, 1(6): 6r-14r.
[6] Lim M Y, You H J, Yoon H S, et al. The effect of heritability and host genetics on the gut microbiota and metabolic syndrome[J]. Gut. 2017, 66(6): 1031-1038.
[7] Dominguez-Bello M G, De Jesus-Laboy K M, Shen N, et al. Partial restoration of the microbiota of cesarean-born infants via vaginal microbial transfer[J]. Nature Medicine. 2016, 22(3): 250-253.
[8] Boix-Amorós A, Collado M C, Mira A. Relationship between milk microbiota, Bacterial load, macronutrients, and human cells during lactation[J]. Frontiers in Microbiology. 2016, 7: e57782.
[9] Fitzstevens J L, Smith K C, Hagadorn J I, et al. Systematic review of the human milk microbiota[J]. Nutrition in Clinical Practice. 2017, 32(3): 354-364.
[10] Fujisaka S, Ussar S, Clish C, et al. Antibiotic effects on gut microbiota and metabolism are host dependent[J]. Journal of Clinical Investigation. 2016, 126(12): 4430-4443.
[11] Xu X, Zhang X. Effects of cyclophosphamide on immune system and gut microbiota in mice[J]. Microbiological Research. 2015, 171: 97-106.
[12] David L A, Maurice C F, Carmody R N, et al. Diet rapidly and reproducibly alters the human gut microbiome[J]. Nature. 2014, 505(7484): 559-563.
[13] Org E, Mehrabian M, Parks B W, et al. Sex differences and hormonal effects on gut microbiota composition in mice[J]. Gut Microbes. 2016, 7(4): 313-322.
[14] Haro C, Rangel-Zuniga O A, Alcala-Diaz J F, et al. Intestinal microbiota is influenced by gender and body mass index[J]. PLoS One. 2016, 11(5): e154090.
[15] Sinha T, Vich V A, Garmaeva S, et al. Analysis of 1135 gut metagenomes identifies sex-specific resistome profiles[J]. Gut Microbes. 2018: 1-9.
[16] Kim Y S, Unno T, Kim B, et al. Sex Differences in Gut Microbiota[J]. The World Journal of Men's Health. 2019, 37: e15.
[17] Bolnick D I, Snowberg L K, Hirsch P E, et al. Individual diet has sex-dependent effects on vertebrate gut microbiota[J]. Nature Communications. 2014, 5(1).
[18] Lee S M, Kim N, Yoon H, et al. Microbial changes and host response in F344 rat colon depending on sex and age following a high-fat diet[J]. Frontiers in Microbiology. 2018, 9: 2236.
[19] Bokulich N A, Subramanian S, Faith J J, et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods. 2013, 10(1): 57-59.
[20] Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics. 2011, 27(16): 2194-2200.
[21] Wang Q, Garrity G M, Tiedje J M, et al. Naive bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology. 2007, 73(16): 5261-5267.
[22] Lozupone C, Knight R. UniFrac: a new phylogenetic method for comparing microbial communities[J]. Applied and Environmental Microbiology. 2005, 71(12): 8228-8235.
[23] Kovacs A, Ben-Jacob N, Tayem H, et al. Genotype Is a Stronger Determinant than Sex of the Mouse Gut Microbiota[J]. Microbial Ecology. 2011, 61(2): 423-428.
[24] Christophe Lay, Lionel Rigottier-Gois, Kim Holmstr?m, et al. Colonic microbiota signatures across five Northern European countries[J]. Applied and Environmental Microboiology. 2005, 71: 4153-4155.
[25] Carmody R, Gerber G, Jr J L, et al. Diet dominates host genotype in shaping the murine gut microbiota[J]. Cell Host & Microbe. 2015, 17(1): 72-84.
[26] Shin J, Park Y, Sim M, et al. Serum level of sex steroid hormone is associated with diversity and profiles of human[J]. Research in Research in Microbiology. 2019: S923-S2508.
[27] Gao X, Zhang M, Xue J, et al. Body mass index differences in the gut microbiota are gender specific[J]. Frontiers in Microbiology. 2018, 9.
[28] Cani P D, Delzenne N M. The role of the gut microbiota in energy metabolism and metabolic disease.[J]. Curr Pharm Des. 2009, 15(13): -.
[29] Pan F, Zhang L, Li M, et al. Predominant gut Lactobacillus murinus strain mediates anti-inflammaging effects in calorie-restricted mice.[J]. Microbiome. 2018, 6(1): 54.
[30] Harley I T W, Giles D A, Pfluger P T, et al. Differential colonization with segmented filamentous bacteria and Lactobacillus murinus do not drive divergent development of diet-induced obesity in C57BL/6 mice[J]. Molecular Metabolism. 2013, 2(3): 171-183.





