摘要:【目的】利用生物信息学的方法,对GEO和TCGA两个基因组学数据库进行分析,探究与前列腺癌相关的差异基因及相关的调控网络。【方法】综合GEO数据库的前列腺癌基因表达芯片数据(GSE46602、GSE55945)和TCGA数据库的RNA-seq数据,利用GEO2R及R语言的edgeR包进行基因差异分析,获得共同的显着差异基因,结合R语言的clusterProfiler包进行GO功能分析及KEGG通路分析,同时利用string网站进行蛋白互作网络分析,筛选出前列腺癌中调节蛋白表达量的关键基因,再结合TCGA临床随访数据分析关键节点基因的临床预后价值。【结果】获得共同差异基因共278个,其中表达上调100个,表达下调178个,它们与上皮细胞的调节增殖、含苯化合物的代谢过程等功能以及谷胱甘肽代谢和粘着斑等信号通路密切相关。蛋白互作网络分析结果得出3个重点蛋白表达模块以及12个关键节点基因。在这些关键基因中,EDN3、EDNRB和AMACR与前列腺癌患者的生存率密切相关。【结论】通过对前列腺癌基因芯片和RNA-seq数据的生物信息学分析,我们发现EDN3、EDNRB与AMACR很可能在前列腺癌的发生发展过程中发挥重要作用。
关键词:前列腺癌; 生物信息学; GEO; TCGA; 差异基因;
Bioinformatics Analysis of Prostatic Carcinoma Via Big-Data
LI Zhi-biao TANG Fu-cai LU Ze-chao HUANG Wei-na HE Zhao-hui
The Third Clinical College of Guangzhou Medical University Department of Urology,The Eighth Affiliated Hospital,Sun Yat-sen University The First Clinical College of Guangzhou Medical University
Abstract:【Objective】The two databases,GEO(gene expression omnibus,GEO)and TCGA(the cancer genome alas,TCGA),were analyzed using bioinformatics methods to screen differentially expressed genes associated and their related regulatory networks in prostate carcinoma. 【Methods】The prostate carcinoma gene expression chip data(GSE46602,GSE55945)downloaded from the GEO database were integrated into the RNA-seq data of the TCGA database. And the differentially expressed genes analysis was performed using GEO2 R and the edgeR package of R software to extract common significant differentially expressed genes. The clusterProfiler package of R software was used to enrich the GO(gene ontology,GO)function enrichment analysis and KEGG(kyoto encyclopedia of genes and genomes,KEGG)pathway analysis. Differentially expressed genes were further constructed into a protein-protein interaction(PPI)network to screen out key genes for regulatory protein expression in prostate carcinoma. Gene analysis results were combined with TCGA clinical follow-up data to analyze the clinical prognostic value of key node genes. 【Results】A total of278 significant differentially expressed genes were extracted,of which 178 genes were down-regulated and 100 genes were up-regulated. These genes were closely associated with the function and pathway enrichment such as the regulation of proliferation of epithelial cells,metabolism of benzene-containing compounds,the glutathione metabolism,and focal adhesion. The protein-protein interaction network analysis revealed three key protein expression modules and 12 key node genes. Among these key genes,EDN3(endothelin-3),EDNRB(endothelin receptor B)and AMACR(alpha-methylacylcoa racemase)were closely related to the survival rate of prostate cancer patients.【Conclusion】Through bioinformatics analysis of gene chip and RNA-seq data in prostate carcinoma,we found that EDN3,EDNRB and AMACR may play an important role in the occurrence and development of prostate carcinoma.
前列腺癌(prostate cancer,PC)是泌尿系统中的常见肿瘤之一。2019年美国预计有174 650例新诊断前列腺癌病例和31 620例死亡病例,其发病率位居男性恶性肿瘤发病率的首位,患者死亡率高居男性恶性肿瘤的第二位[1]。而在中国,前列腺癌的发病率也在逐年增高,已成为男性最常见的十大肿瘤之一,其死亡率也在随着年龄的增长而上升[2]。前列腺癌起病隐匿,临床发现多为晚期[3]且目前治疗方式主要以手术治疗和雄激素阻断治疗为主,但激素依赖性前列腺癌大部分会恶化发展为去势抵抗性前列腺癌,目前尚不能有效地治疗这种病情[4]。因此,前列腺癌的早期诊断,有助于提升患者的生存率,筛选出前列腺癌的生物标志物对其预防和前期的诊治具有重要的意义。人类肿瘤相关的基因表达汇编(gene expression omnibus,GEO)和癌症基因组图谱(the cancer genome alas,TCGA)是目前最大的两个公共组学数据库,近年来,国内外学者通过生物信息学的方法对这两个数据库的数据进行了多项研究[5,6,7]。生物信息学是目前自然科学的研究重点之一,它以计算机作为工具,从核酸和蛋白质序列进行研究,分析序列当中蕴含的结构功能信息。本研究结合两大数据库的基因数据集,筛选共同的差异表达基因,并进一步进行生物信息学分析,为前列腺癌的发生机制与预后预测提供依据。
1 材料与方法
1.1 材料
以“prostate cancer;prostate carcinoma;pros-tate neoplasms;prostatic neoplasm;cancer of theprostate;prostatic cancer”作为关键词,从GEO数据库(www.ncbi.nlm.nih.gov/geo)中搜索基因芯片数据集,筛选标准:(1)数据集为mRNA基因组芯片;(2)具有癌组织与癌旁组织;(3)以AffymetrixHuman Genome U133 Plus 2.0 Array平台作为研究平台;(4)样本数>20。基于以上筛选标准,基因芯片数据集GSE55945和GSE46602纳入研究。GSE46602有36个癌组织样本,14个癌旁组织样本;GSE55945有13例癌组织样本,8例癌旁组织样本。为了进一步利用不同基因组库的大数据集进行验证,从TCGA数据库(http://carcinomage-nome.nih.gov/)下载前列腺癌的RNA-seq数据及相应的随访数据,剔除TCGA数据库中临床随访数据缺失或不全的样本数据,筛选出495例前列腺癌基因数据与98例癌旁数据及其对应的临床随访数据。
1.2 数据处理与基因筛选
利用GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r/)在线分析网站对GEO数据库中的GSE46602和GSE55945基因芯片进行数据处理,筛选差异基因;同时采用R语言的edgeR包对TCGA数据库下载的RNA-seq数据进行差异表达分析。差异基因的阈值设为log2(fold change)>1,P.adjust<0.05。三个数据集差异分析结果绘制火山图,并采用韦恩图取并集。
1.3 通路与功能富集分析
利用R软件的clusterProfiler包[8,9,10]对上述共同差异基因进行GO(gene ontology,GO)功能分析及KEGG(kyoto encyclopedia of genes and genomes,KEGG)通路分析,以P.adjust<0.05为阈值筛查差异基因的主要富集功能和通路。
1.4 蛋白互作网络分析
将交集的差异基因导入string(https://stringdb.org/)在线分析网站进行蛋白质互作网络分析(protein-protein interaction,PPI),将所得结果导入Cytoscape软件,筛选出关键的蛋白表达模块与关键节点基因。
1.5 生存分析
利用R软件survival包,将1.4中获得的前列腺癌关键节点基因表达值按中位数分为高表达和低表达组,结合TCGA数据库中下载的前列腺癌患者预后随访信息,包括总生存时间和生存状态,对两组绘制K-M生存曲线,并进行log-rank统计检验,分析关键节点基因的预后关系,P<0.05为差异有统计学意义。
2 结果
2.1 前列腺癌差异表达基因的筛选
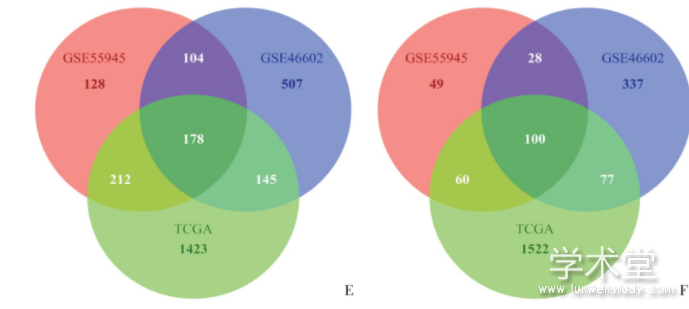
利用GEO2R在线网站对GEO筛选的数据集进行数据处理分析,GSE55945基因芯片筛选结果显示,表达上调的基因622个,表达下调的基因237个;GSE46602基因芯片筛选结果显示,表达上调的基因934个,表达下调的基因542个。利用R软件的edgeR包对TCGA下载的RNA-seq数据进行基因差异表达分析,结果显示,表达上调的基因1 958个,表达下调的基因1 759个。利用R语言的Volcano Plot包做火山图,并利用Venn Diagram包进行基因合集处理(图1)。
2.2 差异基因的通路与功能富集分析
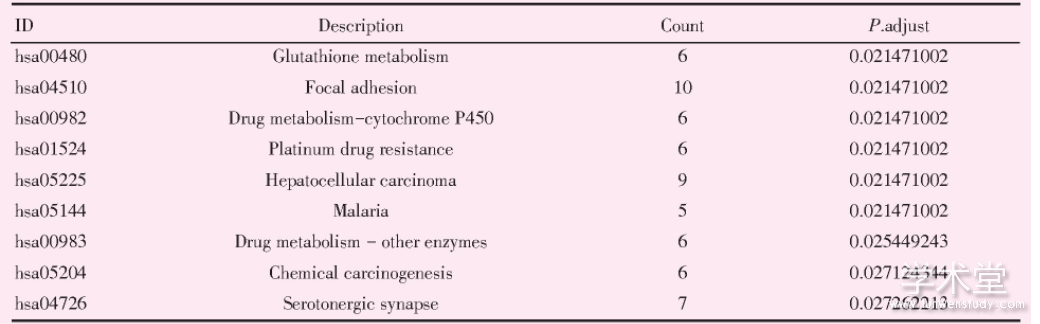
利用R语言的clusterProfiler包对筛选出来的278个共同差异表达基因进行GO功能分析与KEGG通路富集分析,以P.adjust从小到大进行排列,展示GO与KEGG富集分析中的前10富集结果(图2)。结果显示差异基因主要与上皮细胞的增殖和含苯化合物的代谢过程等功能相关(表1),主要富集表达于谷胱甘肽代谢和粘着斑等信号通路(表2)。
表1 前10的GO富集分析结果
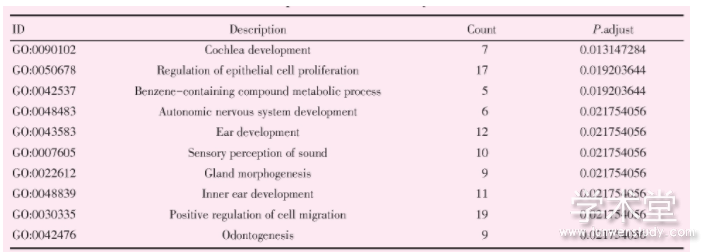
GO:gene ontology.
表2 KEGG通路富集分析结果
KEGG:kyoto encyclopedia of genes and genomes.
图1 差异表达基因火山图和韦恩图
A:Volcano Map of GSE46602 gene chip.B:Volcano Map of GSE55945 gene chip.C:TCGA Database Volcano Map.Red and green spots represent differentially expressed genes,red represent up-regulated genes and green represent down-regulated genes.D:GSE55945,GSE46002gene chip and TCGA data differentially expressed genes Venn diagram.E:GSE55945,GSE46002 gene chip and TCGA data up-regulated gene Venn diagram.F:GSE55945,GSE46002 gene chip and TCGA data down-regulated gene Venn diagram.TCGA:the cancer genome alas.
图2 功能与通路富集分析结果
2.3 蛋白互作网络分析
将交集的差异表达基因导入string在线分析网站进行蛋白互作网络分析,结果进一步导入Cytoscape软件进行PPI网络可视化处理(图3),利用cytoHubba插件对PPI网络中的基因进行节点得分计算,以Degree≥6作为阈值,筛选得到前12个关键基因CAV1、ANXA1、MME、EDNRB、PTGS2、BIRC5、AMACR、PGR、EDN3、GSTP1、CYP3A5及CCK基因,并利用MCODE插件进行重点蛋白表达模块的筛选,以默认参数:Degree≥6,Node Score Cutoff≥0.2,K-core≥2及Max.Depth=100作为筛选标准(图4)。
图3 蛋白质互相作用网络
The red node indicates the up-regulated differentially expressed genes and the green node indicates the down-regulated differentially expressedgenes,and the connected line segments represent mutual regulation between nodes.
图4 重点蛋白表达模块
A:Key protein expression module 1.B:Key protein expression module 2.C:Key protein expression module 3.The red node indicates the up-regulated differentially expressed genes and the green node indicates the down-regulated differentially expressed genes,and the connected line segments represent mutual regulation between nodes.
2.4 生存分析
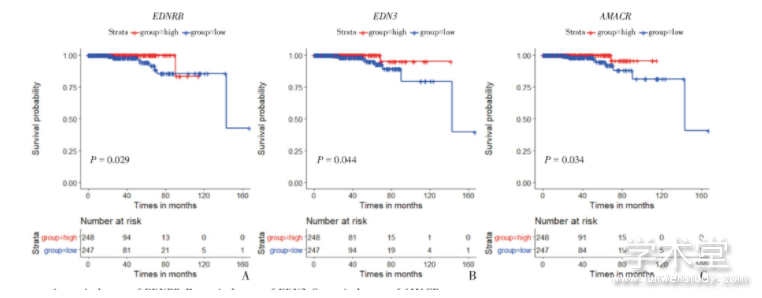
为了探究关键节点基因与前列腺癌的预后关系,利用TCGA随访数据结合关键节点基因表达值进行生存分析,分析结果发现EDNRB、EDN3以及AMACR与前列腺癌预后有紧密联系(P<0.05)。前列腺癌患者中低表达EDN3、EDNRB以及AMACR的患者生存率明显降低(图5)。
图5 生存曲线
A:survival curve of EDNRB.B:survival curve of EDN3.C:survival curve of AMACR.
3 讨论
前列腺癌是泌尿系统好发的癌症之一,全球发病率每年皆呈上升趋势。据文献报道,2015年我国前列腺癌新增人数已超过6万人,死亡人数已达26 600人[2]。前列腺癌有易转移、预后差的特点,因此,筛选前列腺癌的生物标志物对其预防和前期的诊治具有重要的意义,目前,前列腺癌的早期诊治的生物标志物前列腺特异抗原PSA(prostate specific antigen,PSA)已广泛应用于早期筛查中[11]。而在近年来,随着肿瘤分子生物学的发展,基因的靶向治疗也已逐渐得到人们的重视,它具有特异性抗肿瘤的作用,且副作用小,是肿瘤早期筛查与诊治的新技术新方向,具有很好的发展前景[12]。
本研究立足于GEO和TCGA两大组学数据库的基因数据集,采用生物信息学分析方法,对前列腺癌的基因数据进行分析和处理,筛选出共同差异基因278个,差异基因主要汇集于上皮细胞的增殖和含苯化合物的代谢过程等相关功能,并且主要富集于谷胱甘肽代谢和粘着斑等信号通路。同时对交集的差异基因进行PPI网络分析,结果显示CAV1、ANXA1、MME、EDNRB、PTGS2、BIRC5、AMACR、PGR、EDN3、GSTP1、CYP3A5及CCK这12个基因可能在前列腺癌发生的蛋白表达调控机制发挥重要的作用。PPI网络筛选以及生存分析结果显示,EDN3、EDNRB与AMACR很可能在前列腺癌的发生发展及预后中发挥着重要作用。文献报道,目前美国的前列腺癌患者的5年生存率接近100%[13],因此,对前列腺癌远期预后的评估尤为重要。AMACR是CAIB-BALF辅酶A转换酶家族成员之一,编码基因为P504s,是目前前列腺癌诊断的阳性标志物之一[14,15]。Eryilmaz等[16]的结果显示,前列腺癌中AMACR表达明显上调。研究表明,AMACR还可作为前列腺癌的治疗靶点,Festuccia等[17]证明抑制AMACR能有抗前列腺癌的作用,AMACR可作为一个有效的药物作用靶点;Honma等[18]证明AMACR可作为前列腺癌细胞毒性T淋巴细胞免疫疗法的新靶点。本研究发现AMACR在前列腺癌中表达明显上调,与上述文献结果一致,但有关AMACR的表达情况在前列腺癌患者的预后评估未见报道,需要进一步的研究验证。EDN,全称为人内皮素(human endothelin,EDN),据调查,EDN主要由大脑神经细胞、肾小管上皮细胞、前列腺细胞和结肠细胞等细胞分泌得出,主要作用于内皮素受体EDNRA和EDNRB受体[19,20],EDN在多种癌细胞中有着一定的调节作用,在细胞增殖、侵袭以及扩散等过程中起着关键的作用。EDN3与EDNRB的亲和力较高,易结合发挥细胞信号传导的作用[21]。在癌症中,EDNRB可通过促进细胞的凋亡作用及清除ET-1(endothelin-1,ET-1)的效用来拮抗肿瘤的进程[22],文献结果显示,ET-1在癌症中的明显高表达,参与癌症的自分泌和旁分泌途径的调控[23],这表明在前列腺癌中EDN3/EDNRB可能抑制癌症进展。并且文献报道显示,EDNRB在胃癌、结肠癌及前列腺癌具有高甲基化表达的现象,与癌症的进展密切相关,具有重要的研究价值[24,25,26]。上述报道表明EDN3/EDNRB信号通路能对多种癌症的发生进行调控,EDN3与EDNRB均在前列腺癌中低表达,而生存分析结果也显示,EDN3与EDNRB在前列腺癌中发挥着抑癌基因的作用。因此,综合文献挖掘的结果显示,EDN3与EDNRB很可能是前列腺癌的潜在靶基因,对前列腺癌的发生发展及患者的预后评估具有重要的意义,有望成为前列腺癌的早期筛查标记物或基因治疗靶点。
通过对交集的差异表达基因进行通路与功能富集分析结果可知,谷胱甘肽代谢和粘着斑等信号通路与前列腺癌的发生发展机制有密切的联系。研究表明,谷胱甘肽代谢过程中的关键酶谷胱甘肽S-转移酶(glutathione S-transferases,GST)可通过表达沉默参与前列腺癌的发生。Zhao等[27]和Zhou等[28]发现前列腺患者中GSTP1的启动子甲基化发生率明显高于非前列腺癌患者,可能是诊断前列腺癌的潜在生物标记。有学者认为GSTP1的高甲基化可导致其表达沉默,可作为前列腺癌发生早期诊断的重要靶点之一,可调节前列腺癌的发生发展进程[29,30,31],且在多种癌症中都发挥着抑制癌症发展的作用,可用作早期癌症的检测或诊断的生物标志物[32,33]。虽然目前AMACR、EDN3与EDNRB与GST的关系尚未有报道,但我们的研究结果显示,EDN3/EDNRB在肿瘤的作用与其有着一定的相关性,可以有效抑制肿瘤的发展过程且在肿瘤中一般呈高甲基化现象,而AMACR是前列腺癌中的重要标志物,其高表达可能与GSTP1的高甲基化抑制有关,具体机制仍待进一步的实验探索。粘着斑激酶(focal adhension kinase,FAK)与多种癌症的发生发展功能相关,已有研究证明FAK的表达强度与乳腺癌患者的远端转移程度有密切相关[34],在胃癌中FAK信号通路传导的增强也会增强其侵袭能力[35]。据文献报道,FAK通路的激活会导致多种激酶的激活,增强肿瘤的存活和增殖功能[36],而FAK抑制剂有着潜在的抗癌剂的功效[37],文献结果显示,EDN3/EDNRB的抑制也可激活FAK信号通路,增强肿瘤的发生发展进程[38,39]。而AMACR与FAK间的关系尚未见报道,推测AMACR有可能在FAK信号通路的激活上也存在着调控作用,导致癌症的进展加快,但仍需进一步的研究探讨。
综上所述,本研究通过对前列腺癌基因芯片数据集的收集,利用生物信息学分析,筛选出前列腺癌与癌旁组织表达差异的基因,并发现上皮细胞的增殖和含苯化合物的代谢过程等相关功能以及谷胱甘肽代谢和粘着斑等信号通路可能对前列腺癌的发生机制有着密切的关系,同时通过PPI网络分析与生存分析结果表明,EDN3、EDNRB与AMACR作为关键节点基因,很可能在前列腺癌发生发展与预后评估中起重要作用,为前列腺癌发生机制的研究以及新靶点的发现提供理论依据。
参考文献
[1] Siegel RL,Miller KD,Jemal A. Cancer statistics,2019[J]. CACancer J Clin,2019,69(1):7-34.
[2] Chen W,Zheng R,Baade PD,et al. Cancer statistics in China,2015[J]. CACancer J Clin,2016,66(2):115-132.
[3]刘定益,楚晨龙,周燕峰,等. 215例前列腺癌治疗分析[J].中国现代医学杂志,2017,27(8):128-132.Liu DY,Chu CL,Zhou YF,et al. Analysis of 215cases of prostate cancer treatment[J]. Chin J Modern Med,2017,27(8):128-132.
[4] Kim Y,Park SE,Moon JW,et al. Downregulation of androgen receptors by NaAsO2 via inhibition of AKT-NF-κB and HSP90 in castration resistant prostate cancer[J]. The Prostate,2017,77(10):1128-1136.
[5] Wang C,Cai L,Liu J,et al. MicroRNA-30a-5p inhibits the growth of renal cell carcinoma by modulating GRP78 expression[J]. Cell Physiol Biochem,2017,43(6):2405-2419.
[6] Xu S,Kong D,Chen Q,et al. Oncogenic long noncoding RNA landscape in breast cancer[J]. Mol Cancer,2017,16(1):129.
[7] Giulietti M,Occhipinti G,Principato G,et al.Weighted gene co-expression network analysis reveals key genes involved in pancreatic ductal adenocarcinoma development[J]. Cell Oncol,2016,39(4):379-388.
[8] Yu G,He QY. ReactomePA:an R/Bioconductor package for reactome pathway analysis and visualization[J]. Mol Biosyst,2016,12(2):477-479.
[9] Yu G,Wang LG,Han Y,et al. ClusterProfiler:an R package for comparing biological themes among gene clusters[J]. OMICS,2012,16(5):284-287.
[10]Yu G,Wang LG,Yan GR,et al. DOSE:an R/Bioconductor package for disease ontology semantic and enrichment analysis[J]. Bioinformatics,2015,31(4):608-609.
[11]De Nunzio C,Lombardo R,Nacchia A,et al. Repeat prostate-specific antigen test before prostate biopsy:a 20%decrease in prostate specific antigen values is associated with a reduced risk of cancer and particularly of high-grade cancer[J]. BJU Int,2018,122(1):83-88.
[12]Rosenblum D,Joshi N,Tao W,et al. Progress and challenges towards targeted delivery of cancer therapeutics[J]. Nat Commun,2018,9(1):1410.
[13]Allemani C,Matsuda T,Di Carlo V,et al. Global surveillance of trends in cancer survival 2000-14(CONCORD-3):analysis of individual records for37-513-025 patients diagnosed with one of 18 cancers from 322 population-based registries in 71countries[J]. Lancet,2018,391(10125):1023-1075.
[14]Micewicz ED,Khachatoorian R,French SW,et al.Identification of novel small-molecule inhibitors of Zika virus infection[J]. Bioorg Med Chem Lett,2018,28(3):452-458.
[15]Boris P,Gleb S,Nikolay P,et al. Preparation and characterization of the antibody recognizing AMACR inside its catalytic center[J]. Int J Oncol,2018,52(2):547-559.
[16]Eryilmaz IE,Kordan Y,Vuruskan BA,et al. T2E,(TMPRSS2-ERG)fusion transcripts are associated with higher levels of AMACR mRNA and a subsequent prostate cancer diagnosis in patients with atypical small acinar proliferation[J]. Gene,2018,645:69-75.
[17]Zhu J,Sun C,Wang L,et al. Targeting survivin using a combination of miR-494 and survivin shRNA has synergistic effects on the suppression of prostate cancer growth[J]. Mol Med Rep,2016,13(2):1602-1610.
[18]Danilewicz M,Stasikowska-Kanicka O,Wagrowska-Danilewicz M. Augmented immunoexpression of survivin correlates with parameters of aggressiveness in prostate cancer[J]. Pol J Pathol,2015,66(1):44-48.
[19]Takeo M,Lee W,Rabbani P,et al. EdnrB governs regenerative response of melanocyte stem cells by crosstalk with wnt signaling[J]. Cell Rep,2016,15(6):1291-1302.
[20]Watanabe Y,Stanchina L,Lecerf L,et al. Differentiation of mouse enteric nervous system progenitor cells is controlled by Endothelin 3 and requires regulation of Ednrb by SOX10 and ZEB2[J]. Gastroenterology,2017,152(5):1139-1150.
[21]Wang R,Dashwood RH. Endothelins and their receptors in cancer:identification of therapeutic targets[J]. Pharmacol Res,2011,63(6):519-524.
[22]Bagnato A,Loizidou M,Pflug BR,et al. Role of the endothelin axis and its antagonists in the treatment of cancer[J]. Br J Pharmacol,2011,163(2):220-233.
[23]Russignan A,Spina C,Tamassia N,et al. Endothelin-1 receptor blockade as new possible therapeutic approach in multiple myeloma[J]. Br J Haematol,2017,178(5):781-793.
[24]Mousavi Ardehaie R, Hashemzadeh S, Behrouz Sharif S,et al. Aberrant methylated EDNRB can act as a potential diagnostic biomarker in sporadic colorectal cancer while KISS1 is controversial[J].Bioengineered,2017,8(5):555-564.
[25]Tao K,Wu C,Wu K,et al. Quantitative analysis of promoter methylation of the EDNRB gene in gastric cancer[J]. Med Oncol,2012,29(1):107-112.
[26]Yegnasubramanian S,Kowalski J,Gonzalgo ML,et al. Hypermethylation of cpG islands in primary and metastatic human prostate cancer[J]. Cancer Res,2004,64(6):1975-1986.
[27]Zhou X,Jiao D,Dou M,et al. Association of glutathione-S-transferase p1 gene promoter methylation and the incidence of prostate cancer:a systematic review and meta-analysis[J]. J Cancer Res Clin Oncol,2019,145(8):1939-1948.
[28]Zhao F,Olkhov-Mitsel E,Kamdar S,et al. A urine-based DNA methylation assay,ProCUrE,to identify clinically significant prostate cancer[J].Clin Epigenetics,2018,10(1):147.
[29]Sánchez BE,Aguayo A,Martínez B,et al. Using genetic and epigenetic markers to improve differential diagnosis of prostate cancer and benign prostatic hyperplasia by noninvasive methods in Mexican patients[J]. Clin Genitourin Cancer,2018,16(4):e867-e877.
[30]Chatterjee A,Gupta S. The multifaceted role of glutathione S-transferases in cancer[J]. Cancer Lett,2018,433:33-42.
[31]Campos CZ,Losi Guembarovski R,de Oliveira CEC,et al. Glutathione S-transferases deletions may act as prognosis and therapeutic markers in breast cancer[J]. Clin Exp Med,2018,18(1):27-35.
[32]Ogino S,Konishi H,Ichikawa D,et al. Glutathione S-transferase Pi 1 is a valuable predictor for cancer drug resistance in esophageal squamous cell carcinoma[J]. Cancer Sci,2019,110(2):795-804.
[33]Fujikawa Y,Nampo T,Mori M,et al. Fluorescein diacetate(FDA)and its analogue as substrates for Pi-class glutathione S-transferase(GSTP1)and their biological application[J]. Talanta,2018,179:845-852.
[34]Rigiracciolo DC,Santolla MF,Lappano R,et al.Focal adhesion kinase(FAK)activation by estrogens involves GPER in triple-negative breast cancer cells[J]. J Exp Clin Cancer Res,2019,38(1):58.
[35]Wang Z,Wang Z,Li G,et al. CXCL1 from tumorassociated lymphatic endothelial cells drives gastric cancer cell into lymphatic system via activating integrinβ1/FAK/AKT signaling.[J]. Cancer Lett,2017,385:28-38.
[36]Zhang L,Luo B,Dang YW,et al. The clinical significance of endothelin receptor type B in hepatocellular carcinoma and its potential molecular mechanism[J]. Exp Mol Patho,2019,107:141-157.
[37]Guo LL,He ZC,Yang CQ,et al. Epigenetic silencing of olfactomedin-4 enhances gastric cancer cell invasion via activation of focal adhesion kinase signaling[J]. BMB Rep,2015,48(11):630-635.
[38]Rudelius M,Rosenfeldt MT,Leich E,et al. Inhibition of focal adhesion kinase overcomes resistance of mantle cell lymphoma to ibrutinib in the bone marrow microenvironment[J]. Haematologica,2018,103(1):116-125.
[39]Alexopoulou AN,Lees DM,Bodrug N,et al. Focal Adhesion Kinase(FAK)tyrosine 397E mutation restores the vascular leakage defect in endotheliumspecific FAK-kinase dead mice[J]. J Pathol,2017,242(3):358-370.