摘要:目的 · 应用生物信息学方法探讨缺血性心肌病(ischemic cardiomyopathy, ICM)中差异表达的相关基因,构建竞争性内源RNA(competing endogenous RNA, ceRNA)调控网络。方法 · 从基因表达数据库中下载数据集,筛选出ICM样本与正常样本差异表达的mRNA和长链非编码RNA(lncRNA),并进行GO(Gene Ontology)功能分析和KEGG(Kyoto Encyclopedia of Genes and Genomes)通路分析。分析差异表达的m RNA和lncRNA,运用生物信息学方法进行miRNA预测分析,构建ceRNA调控网络。结果· GO功能分析和KEGG通路分析结果表明,差异表达基因在代谢途径、氧化磷酸化、细胞外基质受体相互作用等通路显着富集,并与内质网应激、纤维化、胶原的分解代谢过程、炎症反应功能密切相关。构建了ICM lncRNA相关的ceRNA调控网络,包含26个mRNA、2个lncRNA和15个miRNA。结论 · 采用生物信息学方法能够有效分析ICM差异表达的基因,并成功构建了缺血性心肌病ceRNA调控网络。
关键词:缺血性心肌病; 差异表达基因; 长链非编码RNA; 生物信息学; 竞争性内源RNA;
Bioinformatics analysis of differentially expressed genes in ischemic cardiomyopathy
ZHANG Peng CHANG Zheng-yan YANG Lei XUE Song LIAN Feng
Department of Cardiovascular Surgery, Renji Hospital, Shanghai Jiao Tong University School of Medicine Department of Pathology, Shanghai Tenth People's Hospital of Tongji University
Abstract:Objective · To screen differentially expressed genes in ischemic cardiomyopathy (ICM) by bioinformatics analysis and construct the regulatory network of competing endogenous RNA (ceRNA) . Methods · Data sets were downloaded from Gene Expression Omnibus (GEO) database to screen the differentially expressed mRNAs and lncRNAs between normal samples and ICM ones. Then, GO (Gene Ontology) analysis and KEGG (Kyoto Encyclopedia of Genes and Genomes) pathway analysis were performed. The differentially expressed mRNAs and lncRNAs were analyzed to predict related miRNAs by bioinformatics methods, and then to construct ceRNA regulatory network. Results · GO analysis and KEGG pathways analysis results showed that the differentially expressed genes were enriched in the pathways such as metabolic pathway, oxidative phosphorylation, and extracellular matrix receptor interaction, and they were closely related to the functions such as endoplasmic reticulum stress, fibrosis, collagen catabolic process, and inflammatory response. A ceRNA regulatory network containing 26 mRNAs, 2 lncRNAs and 15 miRNAs was constructed. Conclusion · The bioinformatics method can be used to analyze the differentially expressed genes of ICM, and the regulatory network of ceRNA of ICM has been successfully constructed.
缺血性心肌病(ischemic cardiomyopathy, ICM)是诱发心力衰竭的重要原因。根据美国国立卫生研究院的数据[1,2],ICM是由遗传和环境因素的相互作用引起的。目前,已有多项研究从不同方面对ICM进行了探索。
长链非编码RNA (long non-coding RNA, lncRNA)是一类长度大于200 nt的非编码RNA,是所有非编码转录本的重要组成部分[2]。lncRNA参与机体多个重要的生物学过程[3],也与多个疾病的发生相关[4]。最近研究[5]表明,lncRNA在心血管疾病中发挥重要作用,包括缺血性心肌病;且有研究[6]发现,lncRNA能够扮演竞争性内源RNA (competing endogenous RNA, ceRNA)的角色,通过与mRNA竞争miRNA来调节mRNA的表达。本次研究从基因表达数据库(Gene Expression Omnibus, GEO)中筛选出样本,通过生物信息学方法,分析ICM致病的相关基因,并构建差异表达lncRNA相关的ceRNA调控网络,为ICM治疗探索潜在的靶点。
1 材料和方法
1.1 数据获取
表达谱数据来自美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI) GEO (http://www.ncbi.nlm.nih.gov/geo/)[7],使用数据集系列号为GSE42955(物种:Homo sapiens)。从中筛选ICM样本12例,正常样本(“Normal heart”) 5例。所有样本均采用GPL6244[HuGene-1_0-st]Affymetrix Human Gene 1.0 ST Array[transcript (gene) version]平台检测。
1.2 数据预处理和lncRNA重注释
GEO数据库下载得到cel格式基因表达谱数据,数据利用R软件包oligo[8]采用RMA (robust multiarray average)算法进行背景校正、标准化和汇总。运用芯片平台注释文件,进行重注释。最终得到lnc RNA和m RNA的探针集。通过探针号和m RNA/lnc RNA (gene symbol)的一一匹配,去除没有匹配到的探针;对于不同探针映射到同一基因,取不同探针的均值作为这个m RNA/lnc RNA最终的表达值。
1.3 差异mRNA和差异lncRNA筛选
运用R语言包的limma包,采用经典贝叶斯方法,对ICM样本和正常样本进行m RNA和lnc RNA差异分析,所有m RNA/lnc RNA经过检验后得到相应的P值,将P<0.05的m RNA/lnc RNA视为差异m RNA/lnc RNA。
1.4 差异表达m RNA的功能富集及通路分析
运用富集分析工具DAVID (The Database for Annotation, Visualization and Integrated Discovery, https://david-d.ncifcrf.gov/)[9]对上述差异m RNA进行生物学过程(GO:Gene Ontology, http://geneontology.org/)[10]和通路(KEGG:Kyoto Encyclopedia of Genes and Genomes, https://www.kegg.jp/)[11]分析。显着性阈值P<0.05且富集个数(count)至少为3个的视为显着富集结。
1.5 lncRNA和m RNA协同表达关系预测
运用一一匹配的mRNA和lncRNA数据,计算每个差异mRNA和差异lncRNA的Pearson相关系数,并进行相关性检验,筛选r>0.6且P<0.05的mRNA和lncRNA关系对,认为这些mRNA与lncRNA具有协同表达关系。
1.6 lncRNA功能预测
对预测到的每个lncRNA的靶基因,利用R包clusterProfiler进行GO生物学过程和KEGG通路富集分析,通过富集分析结果来间接预测lncRNA的功能。采用Benjamini-Hochberg方法进行P值矫正,校正后P<0.05且富集个数至少为5个的视为显着富集结果。
1.7 miRNA预测及ceRNA网络构建
对协同表达的差异mRNA使用miRWalk2.0[12]工具,综合miRWalk、miRanDa、miRDB、miRMap、RNA22、Targetscan这6个数据库中的miRNA预测结果,认为在6个数据库的预测结果中均出现的miRNA为调控靶基因的miRNA。对于与差异mRNA协同表达的lncRNA,通过2个工具进行靶向miRNA预测:先利用miRanDa (v3.3a)软件,采用默认参数进行预测,筛选Score≥140、Energy≤-20的miRNA-lncRNA关系对;再利用在线数据库starBase v2.0,预测得到miRNA-lncRNA关系对,取2次预测结果的交集,作为最终的miRNA-lncRNA关系对。综合差异mRNA-miRNA和差异lncRNA-miRNA关系对,首先筛选受到同一个miRNA调控的miRNA-lncRNA-mRNA关系对,结合mRNA与lncRNA的共表达关系(相关系数>0.6),进一步筛选miRNA-lncRNA-mRNA关系对,使用Cytoscape软件进行网络构建,即ce RNA网络。我们认为ceRNA网络中受同一个miRNA调控的具有共表达关系的lncRNA和mRNA互为ceRNA。
2 结果
2.1 差异表达基因的基本信息统计
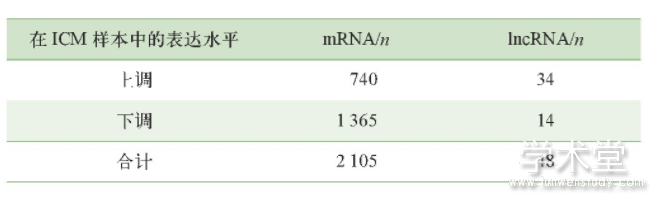
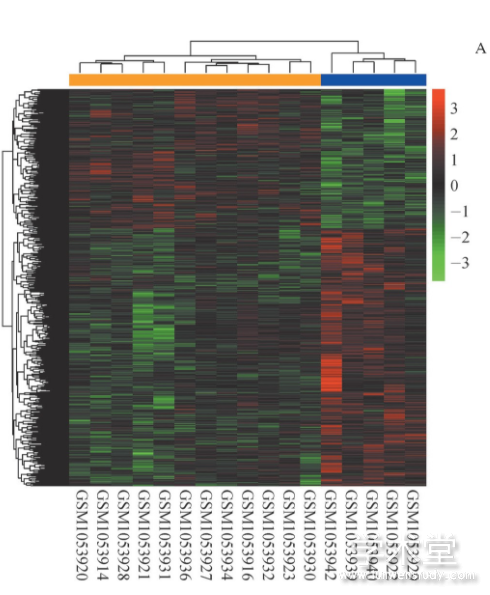
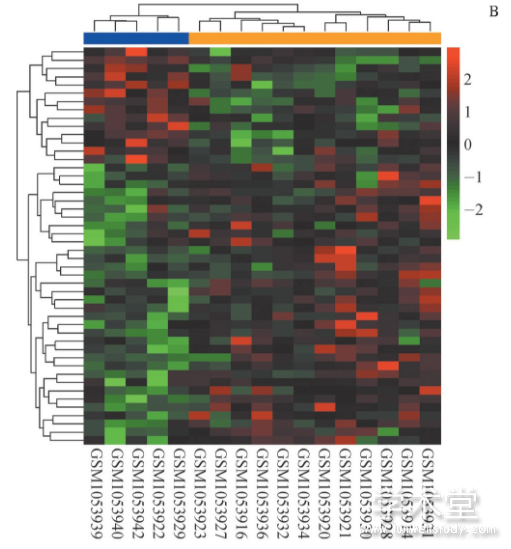
运用芯片平台注释文件共注释了18 392个mRNA和1 384个lncRNA,使用limma包对ICM样本和正常样本分别进行mRNA和lncRNA差异分析,结果如表1所示。基于筛选得到的差异mRNA和差异lncRNA,分别做双向层次聚类热图。如图1所示,差异mRNA和差异lncRNA能够将样本按照预先分组完全分开,说明这些mRNA和lncRNA的改变与ICM的发生有关。
表1 差异m RNA/lncRNA个数统计
图1 差异mRNA和lncRNA的热图
注:A.mRNA的聚类热图;B.lncRNA的聚类热图。顶部蓝色横条表示正常样本,橙色横条表示ICM样本,红色为基因表达水平上调,绿色为基因表达水平下调,颜色深浅表示表达量的变化程度,图片下方为样本编号
2.2 差异mRNA的功能富集及通路分析
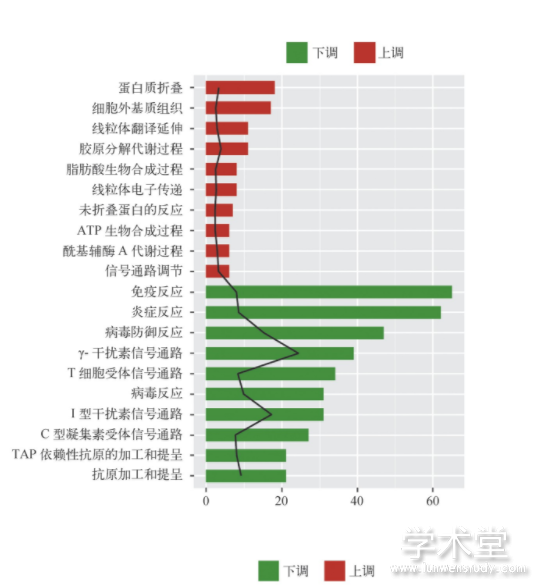
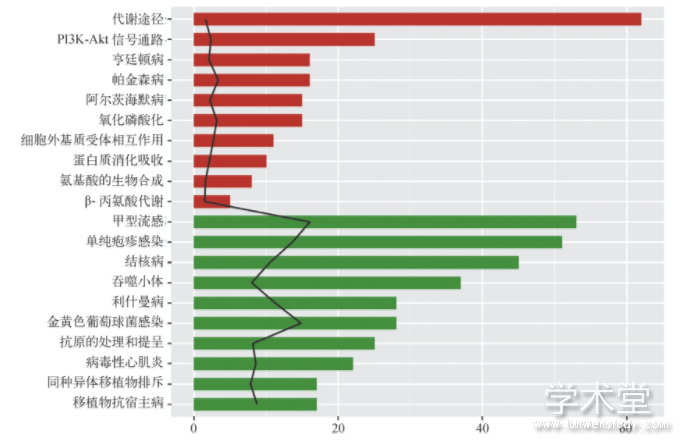
分别对差异分析得到的上调或下调m RNA进行GO功能富集分析和KEGG通路富集分析。上调m RNA显着富集于48个GO生物学过程和12条KEGG通路,下调m RNA显着富集于220个GO生物学过程和66条KEGG通路。如图2所示,按照富集数量排序,上调m RNA在内质网应激(蛋白质折叠和未折叠蛋白反应)、细胞外基质组织、胶原分解代谢等生物学过程,以及代谢途径、氧化磷酸化、细胞外基质受体相互作用等通路显着富集;下调m RNA在炎症反应、免疫反应等生物学过程,以及机体免疫反应信号通路存在显着富集。
图2 差异mRNA的功能分析和通路分析
注:A.GO生物学过程功能分析(上调和下调mRNA富集排名前10个生物学功能);B.KEGG通路分析(上调和下调mRNA富集排名前10条通路)。条带长度表示富集的基因个数;黑色线条表示-lg P
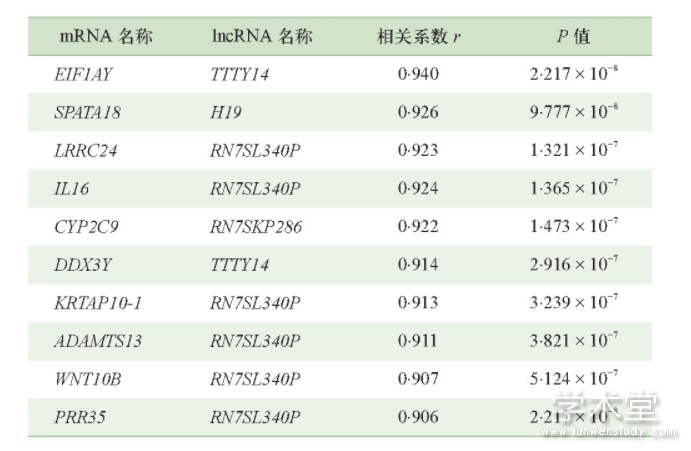
2.3 LncRNA和m RNA协同表达关系预测
共筛选到3 876个协同表达的mRNA-lncRNA关系对,其中包括1 614个m RNA和48个lncRNA。表2统计了相关系数从大到小排名前十的关系对。
2.4 LncRNA功能预测
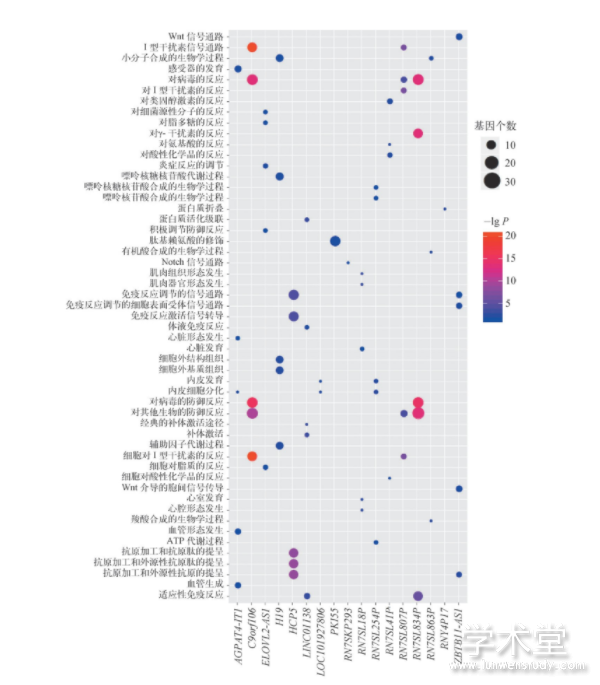
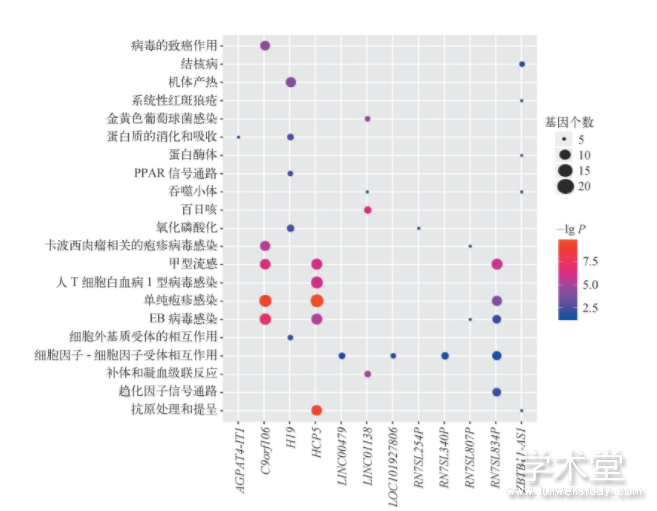
根据预测得到的m RNA-lnc RNA协同关系对,以m RNA作为对应lnc RNA的靶基因,进行GO生物学过程和KEGG通路富集分析,预测对应lnc RNA的功能。每个lnc RNA的GO生物学过程和KEGG通路富集结果排名前五的项目(按照P值排名),如图3、图4所示。共分析出17个lnc RNA的GO生物学过程,12个lnc RNA的KEGG通路。
表2 mRNA-lncRNA关系对的相关性分析
图3 的生物学功能分析结果
注:气泡大小表示富集的基因个数,颜色表示P值的大小
图4 LncRNA的KEGG通路分析结果
注:气泡大小表示富集的基因个数,颜色表示P值的大小
2.5 miRNA预测及ceRNA网络构建
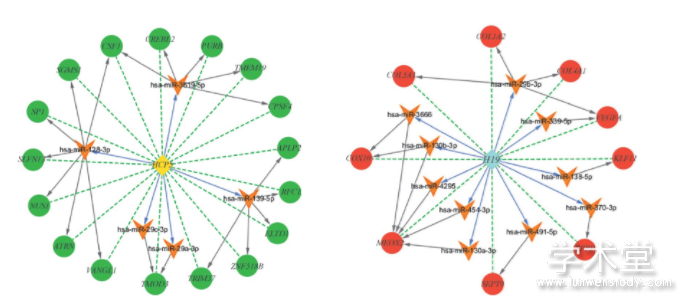
ceRNA网络如图5所示,其中包含34组miRNA-lncRNA-mRNA调控关系,包括15个miRNA、2个lncRNA和26个mRNA。HCP5的下调导致其对hsa-miR-128-3p、hsa-miR-29c-3p、hsa-miR-29a-3p、hsa-miR-139-5p、hsa-miR-3619-5p等miRNA抑制解除,间接抑制了多个mRNA的表达;而H19的上调则导致hsa-miR-29b-3p、hsa-miR-130a-3p、hsa-miR-130b-3p、hsa-miR-138-5p、hsa-miR-339-5p、hsa-miR-370-3p、hsa-miR-454-3p、hsamiR-491-5p、hsa-miR-3666、hsa-miR-4295的表达受到抑制,从而间接调控了多个mRNA的上调。
图5 ceRNA网络图
注:黄色菱形表示下调lncRNA,蓝色菱形表示上调lncRNA;橙色箭头代表miRNA;绿色圆形代表下调mRNA,红色圆形代表上调mRNA;紫色连线代表miRNA-lncRNA调控关系,灰色连线代表miRNA-mRNA调控关系,绿色虚线表示mRNA-lncRNA共表达关系
3 讨论
ICM是冠状动脉粥样硬化终末期病变的一种类型,长期的供氧和需氧不平衡而导致心肌细胞凋亡、坏死,心脏纤维化,瘢痕组织形成,导致预后不佳。长期以来,由于心肌细胞的坏死是一个不可逆转的病理过程,ICM缺乏有效的治疗手段;心脏移植虽能替代受损的心脏,但因供体及免疫排斥的限制尚不能广泛开展。在过去几年中,研究集中在阐明相关蛋白质编码基因和miRNA的分子机制。随着技术的发展,一种包含200多个核苷酸的lncRNA被发现。这种新发现的RNA被证明在广泛的生物学过程中发挥着重要的作用[13]。从癌症到心血管疾病,lncRNA在疾病的发生和发展中起了重要调控作用[14,15]。越来越多的研究[16,17]表明,lncRNA可以靶向结合miRNA,以发挥作为ceRNA的功能,即lncRNA通过竞争性结合miRNA来影响蛋白质编码基因的表达,从而减少蛋白质编码基因的降解。
在本次研究中,我们通过生物信息学方法,对ICM中差异表达的mRNA进行功能注释(GO)和通路分析(KEGG),结果发现其显着富集在纤维化功能和通路以及内质网应激功能和通路中,提示ICM的发病机制与纤维化和内质网应激密切相关。此外,我们还构建了ICM中差异表达的lncRNA相关的ceRNA网络。LncRNA广泛参与了各种病理生理过程的调控,H19属于该家族的重要成员之一,与ICM密切相关。有研究[18]发现,H19在心力衰竭患者左心室组织活检中被发现,而在血管壁中,H19的含量更加丰富;在小鼠心肌肥大模型的心脏组织中也能检测到H19,且其在病理状态下表达上调,并能抑制心肌细胞肥大。
miRNA是一类微小非编码RNA分子,目前的研究已证实miRNA与ICM的发生发展密切相关。Li等[19]的研究结果表明,抑制主动脉狭窄小鼠miR-24的表达可以干预病程的进展;而Xu等[20]通过在心肌细胞过表达miR-24抑制了膜结合蛋白JP2的表达;Wahlquist等[21]则通过抑制miR-25的表达改善了心力衰竭心脏的心肌收缩力。最新的研究[22]发现,miR-21可通过心肌成纤维细胞旁分泌作用于心肌细胞,加剧心肌细胞的肥大。根据本研究构建的ceRNA网络,HCP5与miR-128关系对、H19与miR-29b关系对通过影响多个基因的表达,进而可能对ICM的发生发展产生影响;实验研究也已经证实,miR-128缺失能促进心肌细胞增殖和心脏再生[23],而miR-29b在促进心脏成纤维细胞分泌细胞外基质过程中具有重要作用[24]。在针对膀胱癌细胞的研究中,Lv等[25]发现H19与miR-29b的拮抗关系可影响其侵袭和转移能力,这也与我们所预测的H19与miR-29b之间的关系相吻合。
mRNA即信使RNA,是一大类RNA分子,携带遗传信息,指导蛋白质合成。本次研究通过生物信息学方法构建的ceRNA网络中包含了多个已知的与心血管疾病关系密切的蛋白基因,如α2-Ⅰ型胶原(collagenⅠ-α2, COL1A2)、Kruppel样转录因子11 (Kruppel like factors11, KLF11)、血管内皮生长因子(vascular endothelial growth factor, VEGF)等。COL1A2是Ⅰ型胶原的重要组成部分,Ⅰ型胶原是细胞外基质的主要成分,也是心脏纤维化病变细胞外基质蓄积的主要成分,参与心脏纤维化过程,与ICM的发生发展密切相关[26]。KLF家族是一类在哺乳动物体内广泛分布的锌指蛋白;有研究[27]证实,在小鼠体内过表达的KLF11能抑制心肌肥大和心脏纤维化。VEGF具有特异性促进血管内皮细胞迁移、增殖的作用,可导致血管通透性增加、细胞外基质变性和血管形成等作用,广泛参与了血管的生成;Westenbrink等[28]在研究中指出,VEGF对于促红细胞生成素诱导的心脏功能改善至关重要,促红细胞生成素主要在心肌细胞中促进VEGF表达,并能刺激心肌内皮细胞的增殖。
本次研究主要基于生物信息学分析,未进行实验验证。由于机体的复杂性以及各数据库的局限性,使得某些数据的解释可能会存在一定不足。通过生物信息学方法,我们发现ICM的发病与内质网应激和纤维化的发生密切相关,而lncRNA相关的ceRNA调控网络,对于ICM的诊断、治疗和预后评估具有一定的价值,有利于对疾病的发生发展机制做进一步研究。
参考文献
[1]Rosello-Lleti E, Carnicer R, Tarazon E, et al.Human ischemic cardiomyopathy shows cardiac NOS1 translocation and its increased levels are related to left ventricular performance[J].Sci Rep, 2016, 6:24060.
[2]Herrer I, Rosello-Lleti E, Ortega A, et al.Gene expression network analysis reveals new transcriptional regulators as novel factors in human ischemic cardiomyopathy[J].BMC Med Genomics, 2015, 8:14.
[3]Quinn JJ, Chang HY.Unique features of long non-coding RNA biogenesis and function[J].Nat Rev Genet, 2016, 17 (1) :47-62.
[4]Maass PG, Luft FC, Bahring S.Long non-coding RNA in health and disease[J].J Mol Med (Berl) , 2014, 92 (4) :337-346.
[5]Uchida S, Dimmeler S.Long noncoding RNAs in cardiovascular diseases[J].Circ Res, 2015, 116 (4) :737-750.
[6]Tay Y, Rinn J, Pandolfi PP.The multilayered complexity of ceRNA crosstalk and competition[J].Nature, 2014, 505 (7483) :344-352.
[7]Barrett T, Wilhite SE, Ledoux P, et al.NCBI GEO:archive for functional genomics data sets-update[J].Nucleic Acids Res, 2013, 41 (Database issue) :D991-D995.
[8]Carvalho BS, Irizarry RA.A framework for oligonucleotide microarray preprocessing[J].Bioinformatics, 2010, 26 (19) :2363-2367.
[9]Huang da W, Sherman BT, Lempicki RA.Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J].Nat Protoc, 2009, 4 (1) :44-57.
[10]Ashburner M, Ball CA, Blake JA, et al.Gene Ontology:tool for the unification of biology[J].Nat Genet, 2000, 25 (1) :25-29.
[11]Kanehisa M, Furumichi M, Tanabe M, et al.KEGG:new perspectives on genomes, pathways, diseases and drugs[J].Nucleic Acids Res, 2017, 45 (D1) :D353-D361.
[12]Dweep H, Gretz N.miRWalk2.0:a comprehensive atlas of microRNA-target interactions[J].Nat Methods, 2015, 12 (8) :697.
[13]Engreitz JM, Ollikainen N, Guttman M.Long non-coding RNAs:spatial amplifiers that control nuclear structure and gene expression[J].Nat Rev Mol Cell Biol, 2016, 17 (12) :756-770.
[14]Du Z, Sun T, Hacisuleyman E, et al.Integrative analyses reveal a long noncoding RNA-mediated sponge regulatory network in prostate cancer[J].Nat Commun, 2016, 7:10982.
[15]Greco S, Zaccagnini G, Perfetti A, et al.Long noncoding RNA dysregulation in ischemic heart failure[J].J Transl Med, 2016, 14 (1) :183.
[16]Wang K, Liu F, Zhou LY, et al.The long noncoding RNA CHRF regulates cardiac hypertrophy by targeting miR-489[J].Circ Res, 2014, 114 (9) :1377-1388.
[17]Zhao X, Su L, He X, et al.Long noncoding RNA CA7-4 promotes autophagy and apoptosis via sponging MIR877-3P and MIR5680 in high glucose-induced vascular endothelial cells[J].Autophagy, 2019.DOI:10.1080/15548627.2019.1598750.
[18]Liu L, An X, Li Z, et al.The H19 long noncoding RNA is a novel negative regulator of cardiomyocyte hypertrophy[J].Cardiovasc Res, 2016, 111 (1) :56-65.
[19]Li RC, Tao J, Guo YB, et al.In vivo suppression of microRNA-24 prevents the transition toward decompensated hypertrophy in aortic-constricted mice[J].Circ Res, 2013, 112 (4) :601-605.
[20]Xu M, Wu HD, Li RC, et al.MiR-24 regulates junctophilin-2 expression in cardiomyocytes[J].Circ Res, 2012, 111 (7) :837-841.
[21]Wahlquist C, Jeong D, Rojas-Munoz A, et al.Inhibition of miR-25 improves cardiac contractility in the failing heart[J].Nature, 2014, 508 (7497) :531-535.
[22]Indolfi C, Curcio A.Stargazing microRNA maps a new miR-21 star for cardiac hypertrophy[J].J Clin Invest, 2014, 124 (5) :1896-1898.
[23]Huang W, Feng Y, Liang J, et al.Loss of microRNA-128 promotes cardiomyocyte proliferation and heart regeneration[J].Nat Commun, 2018, 9 (1) :700.
[24]Abonnenc M, Nabeebaccus AA, Mayr U, et al.Extracellular matrix secretion by cardiac fibroblasts:role of microRNA-29b and microRNA-30c[J].Circ Res, 2013, 113 (10) :1138-1147.
[25]Lv M, Zhong Z, Huang M, et al.LncRNA H19 regulates epithelialmesenchymal transition and metastasis of bladder cancer by miR-29b-3p as competing endogenous RNA[J].Biochim Biophys Acta Mol Cell Res, 2017, 1864 (10) :1887-1899.
[26]Khalil H, Kanisicak O, Prasad V, et al.Fibroblast-specific TGF-β-Smad2/3signaling underlies cardiac fibrosis[J].J Clin Invest, 2017, 127 (10) :3770-3383.
[27]Zheng Y, Kong Y, Li F.Kruppel-like transcription factor 11 (KLF11) overexpression inhibits cardiac hypertrophy and fibrosis in mice[J].Biochem Biophys Res Commun, 2014, 443 (2) :683-688.
[28]Westenbrink BD, Ruifrok WP, Voors AA, et al.Vascular endothelial growth factor is crucial for erythropoietin-induced improvement of cardiac function in heart failure[J].Cardiovasc Res, 2010, 87 (1) :30-39.