摘 要: 由于CRISPR(clustered regularly interspaced short palindromic repeats)系统具有多功能基因编辑工具,研究人员能更精确、简便地编辑特定基因组元件.CRISPR技术可对特定位点的DNA或RNA序列进行识别和切割.由于DNA链断裂会启动细胞修复程序,结合CRISPR技术导致靶位点处可设计的基因插入、敲除或替换等.对CRISPR系统的分类进行简介,对CRISPR技术在医学、农业、畜牧领域及高通量筛选领域中的应用进行综述.
关键词: CRISPR;医学;农业;畜牧业;高通量筛选;
Abstract: Researchers can edit specific genomic components more accurately and easily as the CRISPR(clustered regularly interspaced short palindromic repeats) system has a multi-function gene editing tool. CRISPR technology identifies and cleaves DNA or RNA sequences at specific sites. The break of DNA strand initiate cell repair procedures, and the combination with CRISPR technology will lead to editable gene insertion, knockout or replacement at the target site. This paper reviews the classification of CRISPR systems and the application of this technology in the fields of medicine, agriculture, animal husbandry and high-throughput screening.
Keyword: CRISPR; medicine; agriculture; animal husbandry; high-throughput screens;
成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats, CRISPR)由Jansen R等命名[1],其中,CRISPR-associated(Cas)基因总是位于CRISPR基因座附近,表明两者可能具有相互作用关系.2007年,Rodolphe Barrangou等证实,CRISPR-Cas广泛存在于细菌和古细菌中的一套抵御病毒和噬菌体入侵的获得性免疫系统[2].CRISPR系统极具多样性,目前可分成2大类,即干扰效应物由多亚基蛋白复合物组成;干扰效应物依赖单个蛋白质[3].根据特征基因的不同,CRISPR系统又可细分(图1a):Ⅰ型系统的特征蛋白是Cas3,它是一种具有核酸酶和解旋酶结构域的蛋白质;在Ⅱ型系统中,特征Cas9基因编码干扰所必需的唯一蛋白质;Ⅲ型系统以Cas10为特征蛋白,且Cas10组装成类似Cascade的干扰复合体;Ⅳ型系统具有Csf1蛋白质,它是一种未被鉴定的蛋白质,被认为是Cascade复合物的一部分[4];Ⅴ型系统的特征蛋白为Cas9单核酸酶,即Cpf1,C2c1或C2c3[5];Ⅵ型系统的特征蛋白为C2c2,它是含有2个HEPN结构域的RNase蛋白[5].Ⅰ,Ⅲ,Ⅳ型系统基于其多亚基效应复合物发挥功能,而Ⅱ,Ⅴ,Ⅵ型系统基于单个效应蛋白发挥功能.图1a展示了以上6种类型的代表性Cas基因座[6].

图1 CRISPR系统的基本分类和应用情况
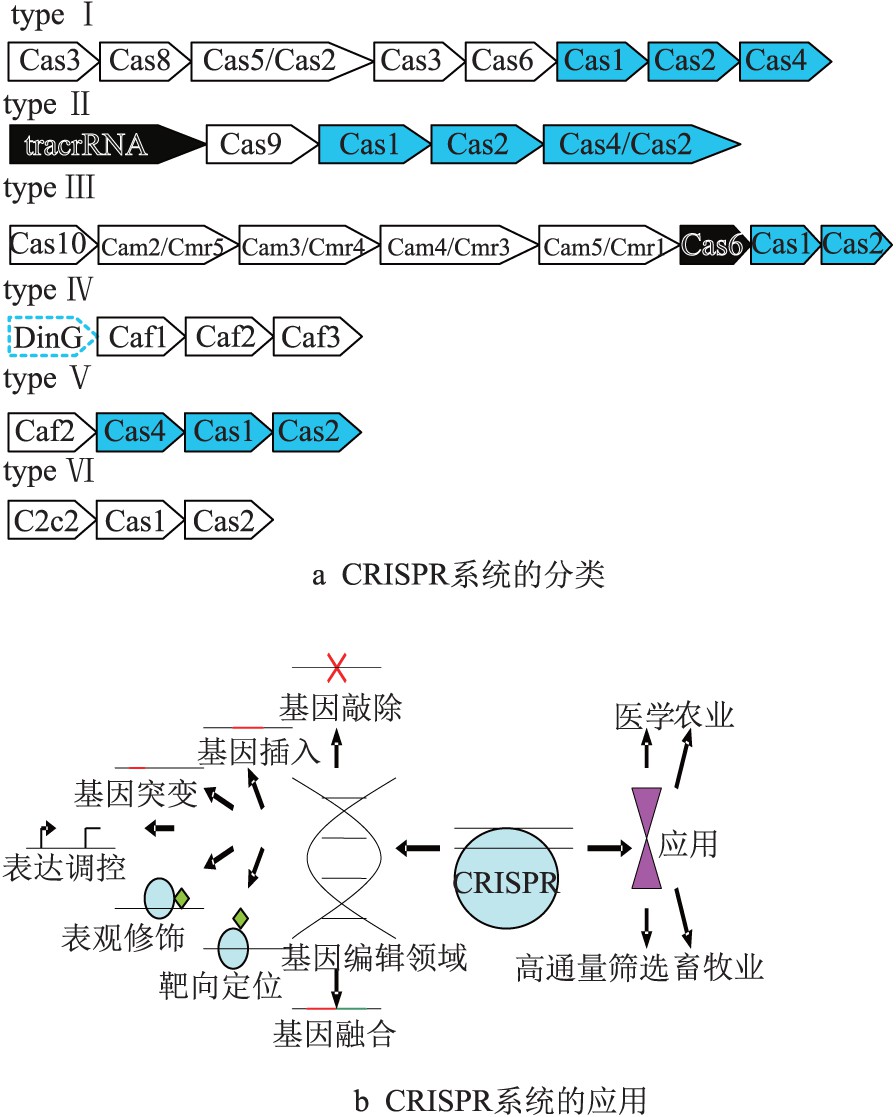
图1中仅存在于某些细菌亚型中的Cas蛋白以虚线轮廓显示,参与干扰的模块为白色,参与crRNA生物发生的模块为黑色,参与适应性的模块为灰色.TypeⅠ中的Cas6同时参与干扰和crRNA生物发生.Ⅳ型系统在没有CRISPR基因座的情况下频繁发生.
由于CRISPR-Cas9系统结构简单,目前它的研究和应用最为广泛.首次发现Cas蛋白可以通过短的RNA引导切割特定的DNA序列后不久[7],Doudna和张锋证实CRISPR介导的基因组修饰可应用于哺乳动物细胞[8,9].目前,CRISPR技术作为新兴的基因编辑工具,与ZFN(锌指核酸酶)和TALEN(转录激活因子样效应物核酸酶)技术一同引领基因工程的新时代[10,11,12].CRISPR技术在基因编辑上的应用大致可分为基因插入[13]、基因敲除[14]、基因定点突变[15]、靶向定位[16]、基因表达调控[17]、基因表观修饰[18]、基因融合[19]等(图1b).在最近的研究中,CRISPR还被应用于选择性消除单条染色体[20]和产生诱导性多能干细胞[21].CRISPR技术不但在实验室基因编辑过程中发挥了重要作用,更在日常饮食、医疗等方面中扮演了越来越重要的角色.笔者综述CRISPR技术在医学、农业、畜牧及高通量筛选等领域的最新进展和应用情况,并对其发展前景进行展望.
1 、CRISPR技术在医学领域的应用
基因治疗是从基因水平上治疗某些疾病的技术.基于DNA或RNA的遗传操纵原理,可将外源正常基因导入靶细胞治疗和预防人类疾病.与传统治疗相比,基因治疗有针对性强、效果显着、患者无痛苦等优势.但也存在安全隐患,面临着稳定性差、效率低、操作复杂的缺陷.CRISPR作为高效、简便、靶向性强的新兴基因编辑工具,对于解决基因治疗的缺陷问题将发挥重要的作用.目前,CRISPR技术在基因治疗领域已经有了广泛的应用,并在病毒性疾病、基因突变性疾病、建立疾病模型、疾病新疗法开发和疾病检测等方面取得了重大进展(表1).
1.1 治疗病毒性疾病
CRISPR作为细菌抵御病毒和噬菌体入侵的免疫系统,可以特异性切割外源基因,目前用于病毒感染类疾病治疗的研究,具体可从靶向切割病毒基因和编辑人体基因组2方面入手:靶向切割病毒基因是在确定病毒的保守序列后,针对其设计gRNA,从而达到清除病毒的目的;编辑人体基因组,则是基于人体细胞自身特性,赋予被编辑的细胞特定性状,或者用于激活免疫系统,以此作为治疗手段.虽然CRISPR技术治疗病毒性疾病应用于临床还有一定的距离,但是其潜力不容忽视.
表1 CRISPR技术在医学领域的应用
2017年, Xu L等通过CRISPR-Cas9系统在造血干细胞移植中靶向切割人CCR5基因[22].将CCR5敲除处理的人HSPC(hematopoietic stem/progenitor cells)重建人免疫系统移植到高度免疫缺陷小鼠后,赋予了小鼠抗1型人类免疫缺陷病毒(human immunodeficiency virus-1, HIV-1)感染的能力.2018年, Ophinni Y等通过CRISPR-Cas9设计6种gRNA[23],其中3种靶向切割HIV-1调节基因Tat,Rev,通过抑制Tat, Rev蛋白的表达,成功减少持续和潜伏感染的CD4+T细胞系中HIV病毒衣壳的产生,可为艾滋病的治疗提供新方法.
高风险人乳头瘤病毒致癌基因(E6,E7)的失调表达是宫颈癌发病和恶化的关键因素.2014年,Zhen S等通过CRISPR-Cas9靶向E6,E7基因的启动子和编码区[24],导致P53,P21蛋白的积累,从而显着降低宫颈癌细胞的体外增殖能力,证明CRISPR技术可以作为宫颈癌和其他HPV相关癌症治疗的新策略.
1.2 、治疗基因突变遗传病
作为一种多用途的基因组编辑工具,CRISPR技术的应用领域十分广泛,如可通过基因定点突变、异常基因靶向敲除等方式应用于基因突变遗传病的治疗[25].其中,单基因突变疾病的治疗研究最为成熟,相信很快可应用于临床.
镰刀型贫血症是一种基因单位点突变疾病,很多科学家已经尝试应用CRISPR技术对其进行治疗[26,27,28].2017年12月,CRISPR Therapeutics公司请求欧洲监管部门允许其开展针对镰状细胞病和β地中海贫血患者的研究性CRISPR治疗方法(CTX001)的临床试验.
Tmc1基因显性突变会引起人和小鼠渐进性听力丧失.2018年,Gao X等通过CRISPR-Cas9基因编辑技术使产生突变的Tmc1基因失活[29],从而改善了小鼠模型中的听力丧失情况.
肌萎缩侧索硬化症(amyotrophic lateral sclerosis, ALS)是一种成人发病的神经系统疾病,患者全身进行性肌肉无力和萎缩,一般发病3~5 a内瘫痪并死亡,目前还没有治愈的方法.SOD1基因的突变常被用于模拟ALS神经变性疾病.2017年, Gaj T等通过CRISPR-Cas9破坏患有肌萎缩侧索硬化症的G93A-SOD1小鼠模型中SOD1的表达[30],与对照动物相比,疾病发作延迟37%,存活率增加25%.
1.3 、建立疾病模型
构建疾病模型对致病机理和治疗方法的研究有重要意义.疾病状态常由单个或多个基因突变导致,在确定导致疾病性状的具体基因和基因突变类型后,研究者可依照所需性状对特定基因进行编辑,以此模拟疾病性状.CRISPR技术具有简便、可遗传和高度稳定等优势,是疾病模型构建领域的有效工具.
杜氏肌营养不良症(duchenne muscular dystrophy, DMD)是由编码抗肌萎缩蛋白的Dmd基因突变引起的X连锁致死性肌肉疾病.2014年,Nakamura K等通过CRISPR-Cas9同时靶向敲除大鼠Dmd基因中的2个外显子[31],结果表明,F0代大鼠中出现抗肌萎缩蛋白表达缺失的性状;Dmd突变的大鼠表现出肌肉力量下降,骨骼肌、心脏和膈肌中退行性/再生性的表型;F1代雄性大鼠的骨骼肌具有相似的表型,证明这些突变可被下一代遗传.从而成功构建了杜氏肌营养不良症的疾病模型.
2017年,Jarno Drost等通过CRISPR-Cas9探索癌症相关突变信号的来源[32],发现错配修复基因MLH1缺陷的类器官中突变积累是由复制错误驱动的,并通过敲除离体器官中的NTHL1基因模拟了在错配修复缺陷的结肠直肠癌中观察到的突变谱.
1.4 、开发疾病新疗法
随着技术的不断改进和融合,CRISPR技术已经被应用于多种新型治疗方法的研究,如与免疫治疗相结合、改变基因表观修饰、寻找药物新靶点等.大量的研究成果让我们感受到CRISPR技术在药物研发和疾病治疗中无限潜能,更让我们对攻克长期危害人类健康的疑难杂症充满信心.
2018年,Kim M Y等在《Cell》上发表了一种能结合CRISPR技术和CAR-T细胞疗法(chimeric antigen receptor T-Cell immunotherapy)治疗急性髓性白血病的新方法[33],即通过CRISPR技术敲除CD33基因,产生白血病特异性抗原,从而赋予CAR-T细胞靶向治疗急性髓性白血病的能力.
2018年,Liu X S等首次通过自主研发的一种移除甲基化的改进型CRISPR-Cas9系统[18],恢复受脆性X染色体综合征影响的神经元中FMR1基因的活性,表明该方法可用于治疗由异常甲基化引起的疾病.
2018年,Barbieri I等构建了多种小鼠白血病细胞模型[34],用于系统性地测试每个可能相关的基因,从而发现哪些基因是AML细胞存活所必需的,最终通过CRISPR-Cas9鉴定出AML白血病的药物新靶标METTL3.
1.5 、检测病毒性疾病
CRISPR家族中的一些成员(Cas12,Cas13)可以靶向切割单链DNA或RNA,研究人员应用这种特性开发出一系列核酸检测新方法.这些核酸检测方法可应用于病毒检测等领域,也为医学检验提供更多选择.
2017年,张锋等开发一种基于CRISPR-Cas13的快速、廉价且高度灵敏的诊断工具SHERLOCK(specific high sensitivity enzymatic reporter UnLOCKing)[35,36],可用于检测RNA或DNA的单分子.出于增强实用性目的,他们又开发出一种微型试纸条测试方法,该方法使肉眼观察测试结果成为可能,且无需使用昂贵的设备.
2018年,Doudna使用被称作DETECTR(DNA endonuclease targeted CRISPR trans reporter)的DNA检测方法实现了灵敏且准确的病毒DNA检测[37],该方法通过在人类样品中检测2种致癌性的人类乳头状瘤病毒得到验证.
2 、CRISPR在农业、畜牧领域的应用
在国家政策的扶持、巨大的市场需求以及相关研究不断取得进展的背景下,CRISPR技术在农业应用方向迎来了新的机遇.与传统技术相比,CRISPR扩展了农业育种工具库,可以更快、更精准地培育出农作物新性状,并极大地缩短育种的时间.同时,使用CRISPR-Cas9技术对农作物基因进行编辑,不涉及外源DNA引入,且经USDA(united states department of agriculture)认定不在转基因监管范围之内,没有食品安全方面的风险.因此,未来CRISPR技术在农业领域更多的突破和发展值得期待.
2.1 、农作物的遗传改良
随着人口的增长,粮食危机成为不得不面对的问题.CRISPR基因编辑技术已经在优化农作物产量、抗病性、口味等性状上取得了一定突破,已成为未来农业育种领域的一大助力.
2.1.1、 提高作物产量
2017年, Zachary B L等通过CRISPR编辑番茄开花抑制因子基因SELF PRUNING 5G,促进番茄的日长敏感性[38],使番茄快速、集中开花,提高了产量.他们还通过CRISPR基因编辑技术成功改良西红柿苗的分枝、花的数量以及果实的大小等性状[39].
氨基酸转运子(amino acid transporters, AATS)在植物发育阶段的营养分配过程中起到不可或缺的作用.2018年, Lu K等通过CRISPR技术抑制氨基酸转运蛋白OsAAP3的表达[40],提高了作物的氨基酸含量,并通过促进水稻的芽生长和分蘖数的增加提高了谷物产量.
2.1.2 、增强抗病性
植物病毒感染不仅影响作物的产量,更对粮食安全构成严重威胁.CRISPR技术应用于作物抗病性研究已有很多实例:2015年,Zahir A等证实CRISPR-Cas9系统可用于植物中以赋予其对DNA病毒的分子免疫[41];2016年,Zhao K等设计的针对水稻OsERF922基因的CRISPR-Cas9 SSN(C-ERF922)[42],改善了稻瘟病抗性;2017年,Peng A等通过CRISPR-Cas9靶向编辑柑橘关键抗性基因CsLOB1启动子[43],大大改善了柑橘的溃疡病抗性.
除此之外,CRISPR系统还可实现对作物抗虫害基因的编辑和筛选,如2018年Khanal C等通过该技术编辑棉花中的肾脏线虫抗性基因[44].
2.1.3 、保障育种安全性
对于食品安全问题,转基因作物备受争议,而CRISPR编辑的作物经USDA认定不在转基因监管范围之内.2016年,Mickael M将纯化的CRISPR-Cas9 核酸酶蛋白直接递送至葡萄和苹果的原生质体内[45],实现非转基因的靶向基因编辑.还有研究者使用农杆菌介导的瞬时CRISPR-Cas9基因表达系统创建非转基因的突变植物[46].这些非转基因的育种方法为育种筛选提供了便利,同时提高了公众的接受度,这对于商业化和大规模生产十分重要.
2.2 、动物育种
自2013年CRISPR-Cas9技术成功应用于哺乳动物细胞研究以来,该技术已经广泛应用于动物育种.
2.2.1、 畜禽遗传育种
健康生活的需求使人们选择食用更多瘦肉,因此提高家畜的瘦肉率是有意义的工作.2017年,赵建国等通过CRISPR向猪细胞内插入UCP1基因[47],该基因负责棕色脂肪组织介导的产热,并在防止感冒和调节能量稳态中起关键作用.UCP1基因的插入可减少脂肪沉积、增加瘦肉率,培育出的猪比正常猪的脂肪少24%,同时抗寒性更好.2018年, Zou Y等通过CRISPR-Cas9进行FBXO40基因的敲除[48],培育出的猪肌肉肥大,且没有可检测的病理效应.
除了改良家畜的肉质品质,CRISPR技术还用于提高家畜的抗病性.禽白血病病毒亚群J(avian leukosis virus subgroup J, ALV-J)已给家禽业造成了重大的经济损失.2017年,Lee H J等通过CRISPR精确编辑鸡细胞中ChNHE1受体基因的关键位点[49],赋予了鸡对ALV-J的抗性,表明该技术可以用于开发抗病品系.
2.2.2 、其他高价值动物遗传育种
CRISPR技术的应用可实现宠物基因靶向编辑[50],满足客户的个性化需求,同时也有助于宠物疾病的治疗.
基因编辑技术还可与克隆技术相结合,通过基因编辑敲除或敲入动物的某个基因,培育出有优良性状的品种,再通过克隆技术大量繁殖.世界首只基因编辑克隆犬“龙龙”在中国诞生,它不仅是一只克隆犬,且其供体细胞来自于世界首例基因编辑疾病模型犬“苹果”.因此,对于有较高价值的动物,通过克隆技术可以扩展基因编辑技术的应用范围.
3 、基于CRISPR技术的高通量筛选
明确产生相应性状的基因是基因编辑技术应用的前提.研究人员通常使用高通量筛选技术确认基因与性状的联系.结合CRIPSR-Cas9和二代测序技术的大规模基因敲除筛选方法,能够极大地推动特异细胞类型及处理条件下筛选相关基因、药物反应及抗药发生等工作,还可快速、准确地找到与某种表型相关的基因,发现新的药物靶标[51].相对于传统的小干扰RNA敲降筛选、常规的突变筛选,CRISPR筛选实验具有通量大、效率高及花费低廉的特点,这些优势使该方法具有巨大的应用潜力.
CRISPR-Cas9文库高通量筛选可以分为阳性筛选与阴性筛选2种模式[52]:阳性筛选对细胞文库施加筛选压力,只有少数目的表型细胞能够存活;阴性筛选中存活的细胞并不是目的表型细胞,因此需要比较不同时间点sgRNA的丰度,找出差异sgRNA确定关键基因(图2).2种筛选方式都常需要与高通量测序技术结合应用[53].
图2 CRISPR-Cas9高通量筛选流程
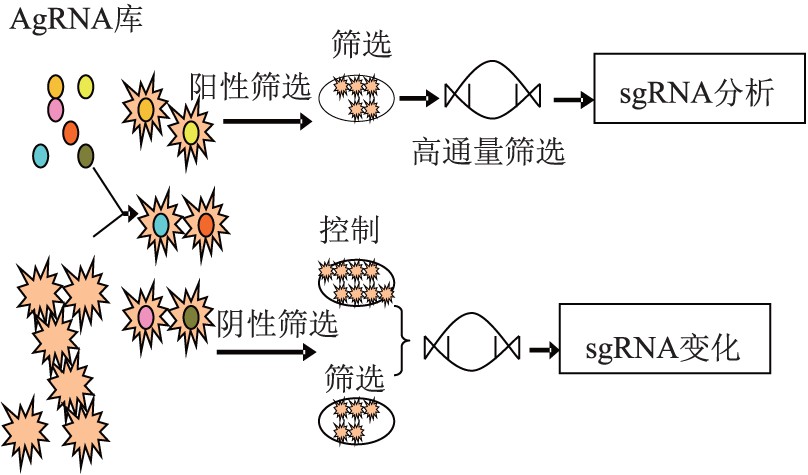
CRIPSR技术应用于高通量筛选已有许多实例,证明其具有实用性和广泛的适用性.2014年,张锋等利用基因组规模的CRISPR-Cas9敲除(GeCKO)文库[54],鉴定癌症和多能干细胞中细胞存活所必需的基因,并在一个黑色素瘤模型中,筛选与vemurafenib耐药性有关的基因,观察到独立的引导RNA对同一基因的高度一致性和高命中率,证明了使用CRISPR-Cas9进行基因尺度筛选的前景.2014年, Zhou Y等通过基因敲除文库筛选[55],成功鉴定炭疽和白喉毒素是细胞中毒所需的宿主基因.
2018年,Pandolfi等开发了一种CRISPR激活lncRNA的筛选策略[56],可靶向14 701种lncRNA基因.他们着重关注控制阿糖胞苷(cytarabine, Ara-C)耐药性的遗传学特征,通过基于CRISPR的高通量筛选方法评估哪些基因可能决定阿糖胞苷的耐药性.CRISPR技术允许研究人员一次分析成千上万个编码基因和lncRNA,并可激活任意感兴趣的基因.2018年,Kurata J S和Lin R J构建了靶向1 594个(85%)已知的人类miRNA茎环的sgRNA库[57],并利用这个sgRNA库筛选影响HeLa或NCI-N87细胞适应度的miRNA.
4 、展望
近年来,CRISPR技术飞速发展,在取得显着成绩的同时也不免引人担忧.最新的研究表明,CRISPR-Cas9基因疗法中发挥作用的细胞通常不携带功能性p53基因,如将这种基因疗法改造过的细胞转移到患者体内,或许会诱发癌症[58].CRISPR技术的脱靶现象也是重要的安全问题,近期《Nature Biotechnology》上的一篇研究报告对CRISPR技术的安全性提出质疑[59],并指出需要进行全面的基因组分析,以便在对患者给药前鉴定具有正常基因组的细胞.以上是CRISPR技术发展中不得不面临的问题,可从提高CRISPR技术安全性和技术性创新两方面着手进行改善.相信经过科学家的不懈努力,CRISPR技术将会给我们带来更多惊喜.
在提高CRISPR技术安全性方面,研究者开发新型Cas9核酸酶提高基因编辑安全性[60].Roth T L等利用电穿孔成功对人T细胞进行CRISPR基因编辑且无需病毒载体[61].Carlson-Stevermer J等利用RNA aptamer分子胶组装出一种CRISPR修复工具包并将其运送到DNA切割位点上[62],大大提高了修复精准度.与传统CRISPR技术相比,该方法的准确率提高了10倍.
在技术创新方面,研究人员也在努力发现新的CRISPR系统,并通过与其他技术结合扩展其应用范围.Silvana Konermann等发现归类为VI-D型的靶向RNA的CRISPR系统[63].受CRISPR系统参与细菌免疫调节机制的启发,Doron S等通过测试抗噬菌体活性[64],发现9个新的抗噬防御系统家族和1个在微生物中广泛存在的抗质粒系统,并显示出强烈的抗外来DNA入侵的能力.一旦它们的机制被破译,将来可能会被改造成有用的分子工具.Hansen-Bruhn M等开发一种使用超声推进的方法将CRISPR-Cas9输送进癌细胞的纳米马达[65],通过可逆的二硫键将Cas9-sgRNA复合物加载到纳米马达表面上,随后超声处理5 min使其穿透细胞质膜,这对临床治疗应用有很大价值.
毫无疑问,未来将会出现更多以新方式应用CRISPR技术来提高人类生存质量的例子.除了利用CRISPR技术独特的优势和能力,研究人员还应该专注于将新的基因编辑工具与其他成熟技术相结合,以提供更灵活、强大的技术手段,从而更好地造福人类.
参考文献
[1] JANSEN R,EMBDEN J D,GAASTRA W,et al.Identification of genes that are associated with DNA repeats in prokaryotes[J].Mol Microbiol,2002 ,43(6):1565-7155.
[2] BARRANGOU R,FREMAUX C,DEVEAU H.et al.CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science,2007 ,315(5819)-1709-1712
[3] LANDER E S.The heroes of CRISPR[J]Cll,.2016, 164(1/2)-:18-28.
[4] MAKAROVA K S,KOONIN E V.Annotation and classification of CRISPR-Cas systems[J]. Methods Mol Biol,2015, 1311:47-75.
[5] SHMAKOV S, ABUDAYYEH 0 O,MAKAROVA K S,et al.Discovery and functional characterization of diverse class 2 CRISPR-Cas systems[J].Mol Cell,2015,60(3):385-97.
[6] WRIGHT A V,NUNEZ J K, DOUDNA J A. Biology and applications of CRISPR systems:Harnessing nature's toolbox for genome engineering[J].Cell,2016,164(1/2):29-44.
[7] JINEK M,CHYLINSKI K,FONFARA I,et al.A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science,2012,337(6096):816-21.
[8] CONG L,RAN F A,COX D,et al.Multiplex genome engineering using CRISPR/Cas systems[J] Science,2013,339(6121):819-823.
[9] JINEK M,EAST A,CHENG A.et al.RNA-programmed genome editing in human cells[J] Elife ,2013,2:e00471.doi: 10.7554/eLife.00471.
[10] PENNISI E.The CRISPR craze[J] .Science ,2013,341(6148):833-836.
[11] KIM Y G,CHA J,CHANDRASEGARAN S .Hybrid restriction enzymes.zinc finger fusions to Fok I cleavage domain[J].Proc Natl Acad Sci USA, 1996,93(3):1156-1160.
[12] REYON D,TSAI S Q,KHAYTER C,et al.FLASH assembly of TAL ENs for high-throughput genome editing[J] Nat Biotechnol,2012 ,30(5):460-465.
[13] AUER T O,DUROURE K,CIAN A D,et al.Highly efficient CRISPR/Cas9-mediated knock-in in zebrafish by homology-independent DNA repair[J].GenomeRes ,2014,24(1):142-153.
[14] BAUER D E,CANVER M C,ORKIN S H.Generation of genomic deletions in mammalian cell lines via CRISPR/Cas9[J].J Vis Exp,2015(95):e52118.
[15] HUANG Xiaosong,WANG Ying,YAN Wei,et al. Production of gene-corrected adult beta globin protein in human erythrocytes differentiated from patient iPSCs after genome editing of the sickle point mutation[J] Stem Cells ,2015 ,33(5):1470-1479.
[16] MA Yuanwu,ZHANG Lianfeng,HUANG Xingxu. Genome modification by CRISPR/Cas9[J]. Febs J,2014,281(23):5186-5193.
[17] LOWDER L G,ZHANG Dengwei,BALTES N J,et al.A CRISPR/Cas9 toolbox for multiplexed plant genome editing and transcriptional regulation[J].PlantPhysiol,2015,169(2):971-985.
[18] LIU X S,WU Hao,KRZISCH M,et al.Rescue of fragile X syndrome neurons by DNA methylation editing of the FMR1 Gene[J].Cell,2018, 172(5):979-992.
[19] MADDAIO D,MANCHADO E,CONCEPCION C P,et al.In vivo engineering of oncogenic chromosomal rearrangements with the CRISPR/Cas9 system[J].Nature, 2014, 516(7531):423-427.
[20] ZUO E,HUO X,YAO X.et al.CRISPR/Cas9-mediated targeted chromosome elimination[J].Genome Biol,2017,18(1):224.
[21] LIU Peng,CHEN Meng,LIU Yanxia,et al.CRISPR-based chromatin remodeling of the endogenous Oct4 or Sox2 locus enables reprogramming to pluripotency[J].Cell Stem Cell,2018,22(2):252-261.
[22] XU Lei,YANG Huan,GAO Yang,et al.CRISPR/Cas9-mediated CCR5 ablation in human hematopoietic stem/progenitor cells confers HIV-1 resistance in Vivo[J]. Mol Ther,2017,25(8):1782-1789.
[23] OPHINNI Y,INOUE M,KOTAKI T,et al.CRISPR/Cas9 system targeting regulatory genes of HIV-1 inhibits viral replication in infected T-cell cultures[J]. SciRep,2018,8(1):7784.
[24] ZHEN S,HUA L,TAKAHASHI Y,et al.In vitro and in vivo growth suppression of human papillomavirus 16-positive cervical cancer cells by CRISPR/Cas9[J].Biochem Biophys Res Commun,2014, 450(4): 1422-1426.
[25] GORI J L,HSU P D,MAEDER M L,et al.Delivery and specificity of CRISPR-Cas9 genome editing technologies for human gene therapy[J].Hum Gene Ther,2015,26(7):443-451.
[26] HOBAN M D,LUMAQUIN D,KUO C Y,et al.CRISPR/Cas9-mediated correction of the sickle mutation in human CD34+ cells[J].Mol Ther,2016,24(9):1561-1569.
[27] YE Lin,WANG Jiaming,TAN Yuting.et al.Genome editing using CRISPR-Cas9 to create the HPFH genotype in HSPCs:.An approach for treating sickle ceII disease and beta-thalassemia[J]. Proc Natl Acad Sci USA,2016,113(38):10661-10665.
[28] BOURZAC K. Gene therapy:Erasing sickle-cell disease[J].Nature,2017.549(7673):s28-s30.
[29] GAO Xue,TAO Yong,LAMAS V,et al.Treatment of autosomal dominant hearing loss by in vivo delivery of genome editing agents[J]. Nature,2018,553(7687):217-221.
[30] GAJ T,OJALA D S,EKMA F K.In vivo genome editing improves motor function and extends survival in a mouse model of AL .S[J]. Science Advances,2017 ,3(12):e3952.
[31] NAKAMURA K,.FUJII W,TSUBOI M,et al.Generation of muscular dystrophy model rats with a CRISPR/Cas system[J].Sci Rep,2014,4:5635.
[32] DROST J,vanBOXTEL R,BIOKZIJL F,et al.Use of CRISPR-modified human stem cell organoids to study the origin of mutational signatures in cancer[J].2017,358(6360):234- 238.
[33] KIM M Y,YU K,KENDERIAN S S,et al.Genetic inactivation of CD33 in hematopoietic stem cells to enable CAR T cell immunotherapy for acute myeloid 1eukemia[J].Cell,2018, 173(6):1439-1453.
[34] BARBIERI I,TZELEPIS K,PANDOLFINI L.et al.Promoter-bound METTL3 maintains myeloid leukaemia by m 6A-dependent translation control[J].Nature,2017.552(7683):126-131.
[35] GOOTENBERG J S,ABUDAYYEh 0 O,KELLNER M J,et al.Multiplexed and portable nucleic acid detection platform with Cas13,Cas12a,and Csm6[J].SCcience ,2018 360(6387):439-444.
[36] GOOTENBERG J S,ABUDAYYEh 0 O,LEE J W,et al.Nucleic acid detection with CRISPR-Cas13a/C2c2[J] Science ,2017,356(6336):438-442.
[37] CHEN J S,MA Enbo,HARRINGTON L B,et al.CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity[J] Science ,2018,360(6387):436-439.
[38] SOYK S,MOLLER N A.PARK S J,et al.Variation in the flowering gene SELF PRUNING 5G promotes day-neutrality and early yield in tomato[J].Nat Genet,2017,49(1):162-168.
[39] SOYK S,LEMMON Z H,OVED M,et al.Bypassing negative epistasis on yield in tomato imposed by a domestication gene[J]Cell,2017, 169(6).1142-1155.
[40] LU Kai,WU Bowen,WANG Jie,et al. Blocking amino acid transporter OSAAP3 improves grain yield by promoting outgrowth buds and increasing tller number in rice[J].Plant Biotechnology Journal,2018,https://doi.org/10. 111/pbi.12907.
[41] ALI Z.ABULFARAJ A,IDRIS A,et al.CRISPR/Cas9-mediated viral interference in plants[J] .Genome Biol,2015, 16:238.
[42] WANG Fujun,WANG Chunlian,LIU Piqing.et al. Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor Gene OsERF922[J].PLoS One,2016,11(4):e0154027.
[43] PENG Aihong,CHEN Shanchun,LEl Tiangang,et al. Engineering canker-resistant plants through CRISPR/Cas9-targeted editing of the susceptibility gene CsLOB1 promoter in citrus[J].Plant Biotechnol J,2017,15(12):1509-1519.
[44] KHANAIL C,MCGAWLEY E C,OVERSTREET C.et al.The elusive search for reniform nematode resistance in cotton[J] Phytopathology,.2018, 108(5):532-41.
[45] MALNOY M,VIOL A R,JUNG M,et al.DNA-free genetically edited grapevine and apple protoplast using CRISPR/Cas9 ribonucleoproteins[J].Front Plant Sci,2016,7:1904.
[46] CHEN Longzheng,LI Wei,KATIN-GRAZZINI L ,et al.A method for the production and expedient screening of CRISPR/Cas9-mediated non-transgenic mutant plants[J].Hortic Res ,2018,5:13.
[47] ZHENG Qiantao,LIN Jun,HUANG Jiaojao,et al.Reconstitution of UCP1 using CRISPR/Cas9 in the white adipose tissue of pigs decreases fat depositionand improves thermogenic capacity[J]. PNAS,2017.114(45):e9474-e9482.
[48] ZOU Yunlong,LI Zhiyuan,ZOU Yunjing.et al.A FBXO40 knockout generated by CRISPR/Cas9 causes muscle hypertrophy in pigs without detectable pathological effects[J]. Biochem Biophys Res Commun,2018, 498(4):940-945.
[49] LEE H J,LEE K Y,JUNG K M,et al.Precise gene editing of chicken Na+/H+ exchange type 1 (ChNHE1) confers resistance to avian leukosis virus subgroup J (ALV-J)[J].Dev Comp Immunol,2017,77:340-349.
[50] Z0U Qingjian,WANG Xiaomin,LIU Yunzhong,et al.Generation of gene -target dogs using CRISPR/Cas9 system[J].J Mol Cell Biol,2015,7(6):580-583.
[51] MANGUSO R T,POPE H W,ZIMMER M D,et al.In vivo CRISPR screening identifies Ptpn2 as a cancer immunotherapy target[R].Nature ,2017 ,547(7664):413-418.
[52] XU Han,XIAO Tengfei,CHEN Chenhao,et al Sequence determinants of improved CRISPR sgRNA design[J] Genome Res,2015,25(8):1147-57.
[53]李欢欢,黄乘浩.基于CRISPR-Cas9的功能基因筛选研究进展[J].生物工程学报, 2018,34(4):461-472.
[54] SHALEM O,SANJANA N E,HARTENIAN E et al. Genome -scale CRISPR-Cas9 knockout screening in human cells[J] Science,2014,343(6166):84-87.
[55] ZHOU Yuexin,ZHU Shiyou,CAl Changzu,et al.High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells[J].Nature ,2014, 509(7501):487-491.
[56] BESTERAC,LEE J D,CHAVEZ A,et al.An Integrated genome-wide CRISPRa approach to functionalize IncRNAS in drug resistance[J]. Cell,2018,173(3):649-664.
[57] KURATA J,LIN R.MicroRNA-focused CRISPR-Cas9 library screen reveals fitness-associated miRNAS[J].RNA.2018,24(7):966-981.
[58] MORGUNOVA E,YIN Yimeng,JOLMAA,et al. Structural insights into the DNA- binding specifity of E2F family transcription factors[J].Nature Communications, ,Doi:10. 1038/ncomms 10050.
[59] KOSICKI M,TOMBERG K,BRADLEY A.Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements[J].Nat Biotechnol,2018,36(8):765-771.
[60] WEBER E R,HELPS C R,FOSTER A P,et al.Molecular cloning and phylogenteic analysis of a CDNA encoding the cat (Felis domesticus) Ig epsilon constant region[J].Veterinary Immunology and Immunopathology,2000.76:299-308 .
[61] ROTH T L,PUIQ-SAUS C,YU R,et al. Reprogramming human T cell function and specificity with non-viral genome targeting[J].Nature. 2018, 559(7714):405-409.
[62] CARLSON-STEVERMER J,ABDEEN A A,KOHLENBERG L,et al.Assembly of CRISPR ribonucleoproteins with biotinylated oligonucleotides via an RNAaptamer for precise gene editing[J].2017,8(1):1711.
[63] KONERMANN S,LOTFY P,BRIDEAU N J,et al.Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors[J].Cell,2018, 173(3):665-676
[64] DORON S,MELAMED S,OFIR G,et al.Systematic discovery of antiphage defense systems in the microbial pangenome[J].Science ,2018 doi:10.1126/Science. aar4120.
[65] HANSEN-BRUHN M,deAVILA B E,et al.Active intracellular delivery of a Cas9/sgRNA complex using ultrasound-propelled nanomotors[J].Angewandte Chemie Internation Edition,2018, 57(10):2657-2661.





