第三章 黄瓜白粉病抗病基因的全基因组关联分析
黄瓜白粉病的抗性机制十分复杂,从之前的 QTL 定位研究结果看,不同的抗病材料所携带的抗病基因不同。同一份抗病材料也可能携带不止一个抗病相关基因,且显隐性也各不相同。如何从广泛的遗传资源中综合全面地挖掘抗病基因一直是一个难以解决的难题,按照之前的研究思路一般是先通过基因定位然后进行基因克隆,但是这种方法只能对个别抗病材料中的抗病基因进行挖掘,难以进行综合研究。随着二代测序技术的发展,全基因组关联分析(GWAS)技术成为了研究复杂性状的一种非常有力的研究工具,基于统计学的相关性分析,可以直接利用自然群体进行基因定位研究。现已在人类和重要的植物模式作物中取得了不少研究成果。本实验利用已构建的黄瓜核心种质,进行 GWAS 研究,在全基因组范围内挖掘黄瓜白粉病抗性基因。
3.1 GWAS 材料方与法
3.1.1 实验材料
本实验所采用的研究材料由中国农科院蔬菜花卉所黄瓜课题组提供的黄瓜115份核心种质材料,由于部分材料缺失,实验实用 109 份材料。这份核心种质是从来自世界各地 3000 多份种质中筛选出来的,详见齐建建(2014)。整个核心种植群体均完成了全基因组的重测序(Qi et al.2013),测序数据见黄瓜基因组核心种质抗病性鉴定。
3.1.2.1 材料准备
鉴定于 2013~2014 年在中国农业科学院蔬菜花卉研究所内和顺义实验基地的温室进行。鉴定实验分 3 次进行:2013 年春,2013 年秋和 2014 年春。按照田间正常管理,进行田间成株期自然发病下的抗病性鉴定。所有株系分三个重复,每个重复种质 6 株。
3.1.2.2 病情调查和数据处理
抗病性鉴定采用的方法是成株期自然发病,所以不需要接种,待所有株系充分发病后,进行病情调查,调查和数据统计方法参照上一章节。不同季度抗病鉴定的结果进行相关性分析,所用的软件为 SAS V9.2。
3.1.3 全基因组关联分析
3.1.3.1 群体结构和连锁不平衡分析
利用 STRUCTURE V2.3.1 进行群体结构分析,由于使用的是相同一份核心种质,详细步骤和结果可参见 Qi et al(2013)。本实验为了简化群体结构复杂性对 GWAS 结果的影响,K 值取为 3。连锁不平衡分析方法也结果也参见 Qi et al(2013),本实验直接使用该研究的结果,做后续分析。
3.1.3.2 黄瓜白粉病抗性关联分析
利用 TASSELV3.0 进行白粉病抗性基因的关联分析,选用一般线性模型(GLM),对 SNP 标记与表型数据进行全基因组关联分析。用 TASSEL 自带的画图功能绘出曼哈顿图。
3.2 GWAS 结果与分析
3.2.1 抗病性鉴定结果
分别于 2013 年春(pm_2013)、2014 年春(pm_2014_s)、2014(pm_2014_a)年秋三个季度对核心种质材料进行了抗白粉病鉴定。并对鉴定结果进行了统计分析(详见图 3.1 和表 3.1)。
从抗病鉴定的结果来看,抗性材料在三个季度中的表现均比较一致,如 CG7、CG9、CG32、CG44、CG101 在三个季度中均表现为高抗,病情指数低于 5,另有 CG70 和 CG82 连续两个季度均表现出完全抗性。感病性状表现稳定的株系包括 CG28、CG62、CG78、CG109、CG120 等,这三个株系的最低病指都大于 45。但是有的中抗或中感的材料表现较不稳定。三个季度的变异系数介于 52.7%和 80.0%之间。
表 3.1 核心种质群体三个季度抗病鉴定病指统计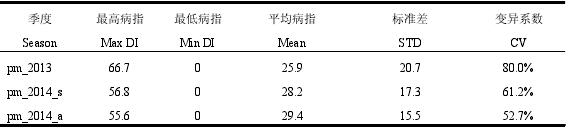
3.2.2 全基因组关联分析
利用核心种质三个季度的抗病性鉴定的结果与 SNP 标记进行全基因组关联分析,并绘制曼哈顿图,如图 3.1。可以发现,有多个信号位点被检测到。关联分析所用的 SNP 数据来源于 Qi et al.(2013)和黄瓜基因组从检测结果来看,在三个季度中共监测到 6 个信号区域,全部出现在 2013 季度实验中。其中 pmG2.1、pmG5.2 和 pmG5.3 在后续两个季度的实验中被重复检出,是表达稳定的抗性位点。然后选取各个信号区间内-log P 值最高的 SNP 的物理位置代表其所在的区段,详见表 3.2.
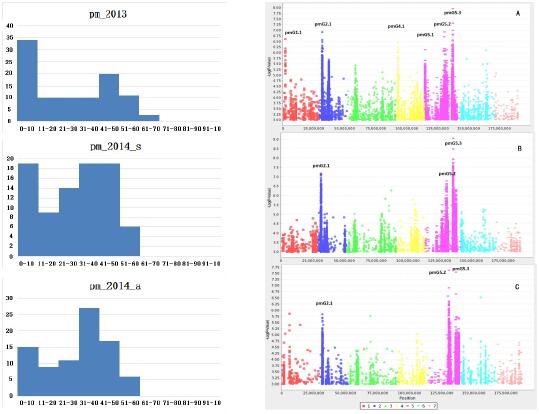
图 3.1:核心种质三个季度抗病鉴定 DI 频数分布和全基因组关联分析曼哈顿图
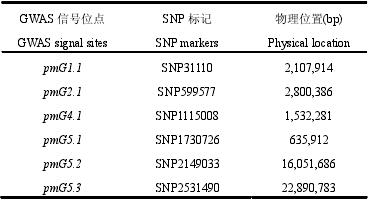
3.2.3 关联分析结果与 QTL 结果比较
将 GWAS 检测到的 6 个信号位点的位置与前人 QTL 定位结果进行了比对,发现三个稳定表达的位点与之前定位的 QTL 位点基本一致。详见表 3.3,另外有 pmG2.1 和 pmG4.1 未找到相对应的 QTL 位点。
表 3.3 GWAS 位点与前人 QTL 定位结果比对
3.3 讨论
本实验通过对黄瓜核心种质进行了三个季度的抗病性鉴定,结合重测序数据进行了全基因组关联分析,最终检测到 6 个信号区域,其中 3 个与前人 QTL 定位结果基本一致,分别为:pmG1.1、pmG5.2、pmG5.3,特别是 pmG5.3 这个位点,不仅是本实验检测到的信号最强的位点,也是前人QTL 定位到的次数最多的一个区域,说明 GWAS 在黄瓜白粉病抗性基因检测中的应用是有非常效并且可靠的。另外的 3 个位点并没有被之前的 QTL 实验检测出来,我们推测可能是因为此具有此位点抗性基因的材料还并未被研究人员所研究。pmG2.1 在三年的 GWAS 分析中都检测到,并且信号很强,但是根据前人的 QTL 分析结果发现,2 号染色体上并未检测到抗性相关区域,可能就属于这种情况。
因此,下一步可以根据 GWAS 结果,采用生物信息学方法的手段,将抗性材料与其对应的抗性位点一一对应,然后通过构建遗传群体等方法,分别进行遗传定位,验证 GWAS 结果的可靠性,深入挖掘白粉病抗性基因。黄瓜白粉病抗性是一种遗传特别复杂的性状,往往涉及到多个基因或多条代谢通路。使用基因定位的手段往往只能定位到对性状影响最显着的几个基因,并且由于不同的抗病材料具有不同的抗病基因,不可能从一个群体或者一份材料中克隆出所有与白粉病抗性相关的基因。但是GWAS 技术的出现解决了这一难题,黄瓜核心种质包含了大多数的遗传变异,代表了最广泛的遗传多样性,可以同时对所有的遗传资源中的抗病基因进行检测,在全基因组范围挖掘抗病基因。GWAS 为全面的研究白粉病抗性机制,提供了一个非常有效的手段。





