摘 要: 本文以浙江某校内河道某河段作为试验区,在种植植物的基质区(A)和无植物的基质区(B)分别于水深20 cm和50 cm处取样,分析2处不同试验体系下人工基质上生物膜的微生物群落结构,采用高通量测序技术,分析微生物的群落组成、丰度和多样性。结果表明,变形菌门、浮霉菌门、拟杆菌门是2组基质上的优势门。在种植植物区的物种丰度为A20>A50,无植物区的B20、B50处变形菌门、浮霉菌门的物种丰度差异不明显;不同采样位点微生物的OTU组成存在明显差异,Shannon及Simpson指数显示,生物多样性差异和均匀度没有显着变化。
关键词: 人工基质; 微生物; 群落结构; 高通量测序;
河流是陆地水生态系统的重要组成部分,相比自然河流,城市河流受长期人为的影响较大[1],污染严重时则会制约经济发展。新型生态护坡基质通过搭配种植植物固定在河岸边,具有生态美观、经济适用、施工简便等特点,对恢复河道的自净能力及对微生态系统的形成具有促进作用,应用前景良好[2]。微生物在河流生态系统氮、磷的转化中发挥重要的作用,因此,对城市河道基质上微生物的研究具有重要的生物学意义,并为生态治理和环境保护提供应用价值[3]。
由于校内河道水流较缓,处于相对静止的状态,能创造一个相对稳定的生境,为一些相关试验的开展提供了良好的野外条件。本研究拟以人工基质上生物膜为研究对象,通过刮取基质上的生物膜得到待测样品,采样高通量测序技术分析不同环境条件下微生物的群落结构差异。高通量可以通过对16S rDNA、18S rDNA、ITS区域进行扩增测序[4],16S rDNA为编码原核生物核糖体小亚基rRNA的DNA序列,具有10个保守区域和9个高变区域(V1~V9)。保守区在细菌中差异不大,高变区具有属和种的特性。通过对某一段高变区序列(V4区或V3~V4区)进行PCR扩增后进行测序[5]。不同的16S rDNA序列的相似性高于97%就可将其归属为一个操作分类单位(operational taxonomic unit,OTU),每个OTU近似对应于物种水平[6]。以16S rDNA扩增进行测序分析主要用于微生物群落多样性和构成的分析,大大拓展了对环境微生物的微生态认知。通过研究2种不同环境下护岸基质上的微生物群落结构的多样性,确定了优势类群,可为河道特定功能菌群的进一步研究和环境对微生物的影响提供参考依据。
1 、材料与方法
1.1、 试验材料
本试验采用的材料为聚氯乙烯或聚丙烯经乱丝热熔相互搭接,再经模具挤压成型的三维网状基质。
1.2、 样品采集
采样区域位于温州医科大学茶山校区一段河道内,根据采样点的布设,分别从种植植物的基质上(A)和未种植植物的基质上(B)刮取生物膜样品,样品在每个基质距水面20和50 cm处刮取。样品采集时间为2018年5月18日,用已灭菌的刀片和刷子刮取生物膜,置于已灭菌的2 mL的离心管中,尽快带回实验室中,于-80 ℃保存待测定。

1.3、 高通量测序分析
样品委托上海祥音生物科技有限公司利用Illumina HiSeq 2500进行高通量测序。通过Barcode区分样品序列,并对各样本序列做质量控制,去除非特异性扩增序列及嵌合体。在97%的相似性水平下,基于OTU聚类,即获得各OTU的物种分类信息,基于此对每个OTU进行物种分类和汇总,得出丰度数据表,再根据Chao1丰富度估计量,Shannon指数和Simpson指数,进而判断生物群落的结构特点。数据整理采用Excel 2016,使用Origin 8.1软件进行绘图处理。进行各样本多样性指数、群落结构及种群相对丰度差异分析。
2 、结果与分析
2.1、 多样性
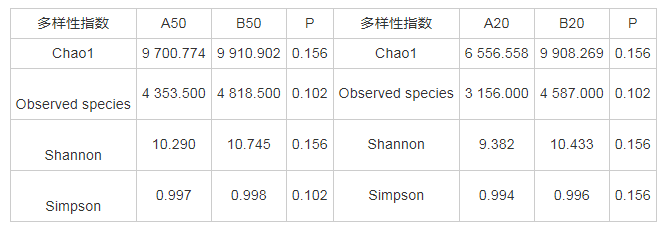
Alpha多样性反映的是单个样品内部的物种多样性,包括observed species、Chao1,Shannon及Simpson指数等。其中,OTU数为A50:12 143,A20:8 681,B50:11 919,B20:14 458。各样品的OTU数存在较大差异,其中B20的OTU数量最高。通过observed_species和Chao1指数可以估计富集样品的群落丰度,通过Shannon和Simpson指数可以估计样品的群落多样性。由表1可知,4个位点样品的群落多样性比较相近,而群落丰度上差异较大。
表1 不同位点纤维基质上物种的α多样性指数
2.2 、群落组成
各采样点样品中所含的微生物涉及21个门21个纲,并存在不少OTU归为Unclassified。各样品优势门水平(相对丰度超过1%)的菌群组成情况见图1。
图1 不同位点基质上的物种在门水平上群落结构和分布
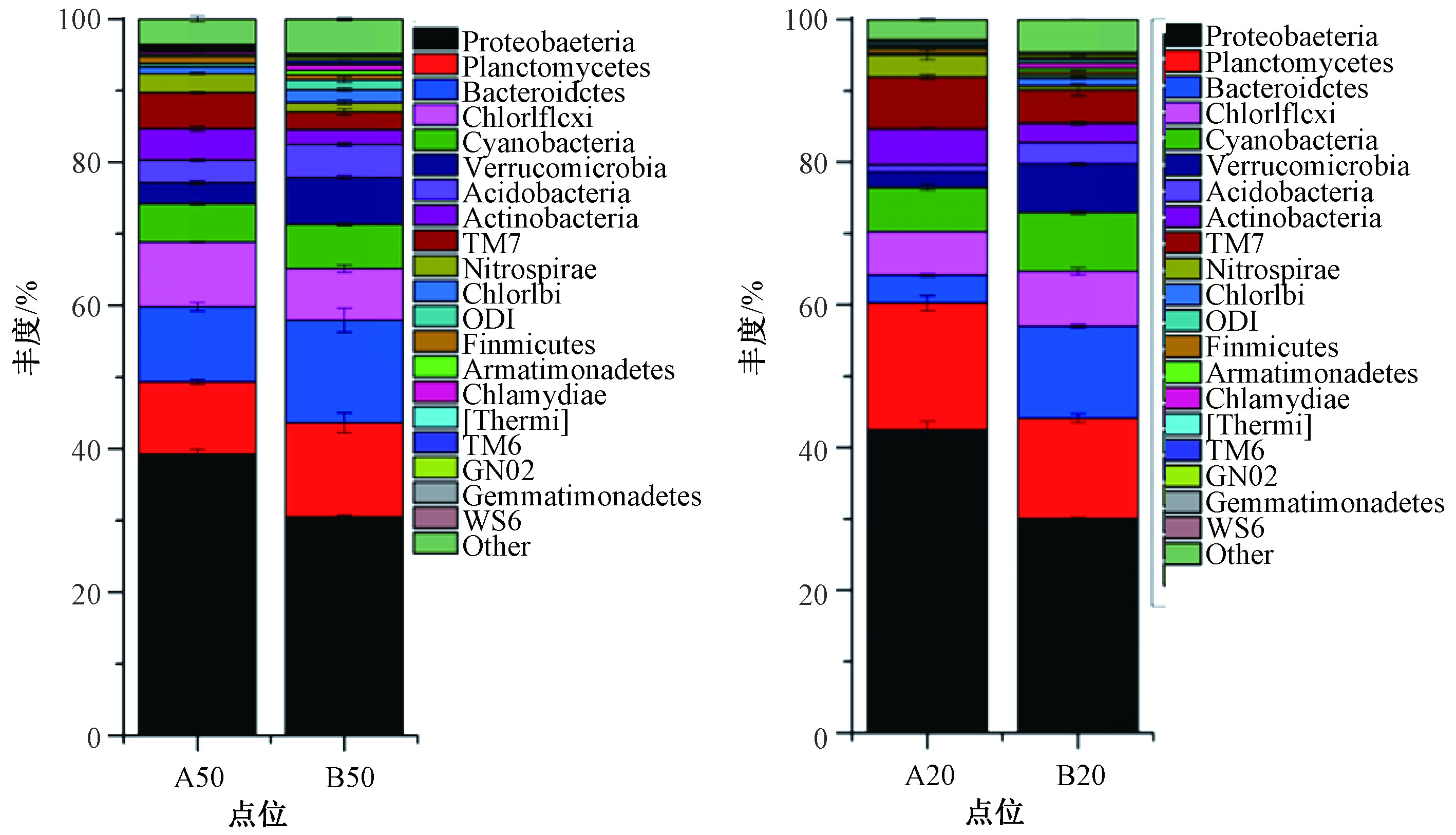
由图1可知,取相对丰度高于0.1%的物种作为优势物种,其中变形菌门(Proteobacteria)、浮霉菌门(Planctomycetes)、拟杆菌门(Bacteroidetes)和蓝藻细菌(Cyanobacteria)在各位点中所占比例最为明显,为样品中的优势门,其中变形菌门具有最高的相对丰度,在A区的变形菌门的相对丰度达到39.3%左右,B区变形菌门相对丰度较A处低,大致在30.5%。
在纤维基质上,不同试验位点存在显着差异。在深层和浅层的纤维基质上附着的变形菌门(Proteobacteria)在2个采样点变化趋势基本一致,其中在A区,纤维基质上的变形菌门(Proteobacteria)大于B区,说明植物的搭配种植,提高了变形菌门(Proteobacteria)的生存环境,提高了其丰度。说明变形菌门(Proteobacteria)可能依赖于植物生长提供的环境,或者说有植物的基质能有利于变形菌门(Proteobacteria)的生长。
在纤维基质上不同试验位点上存在显着差异,在深层和浅层的纤维基质上附着的变形菌门在2个采样点变化趋势基本一致,其中在A区,纤维基质上的变形菌门大于B区,说明植物的搭配种植,提高了变形菌门的生存环境,提高了其丰度。说明变形菌门可能依赖于植物生长提供的环境,或者说有植物的基质能有利于变形菌门的生长。
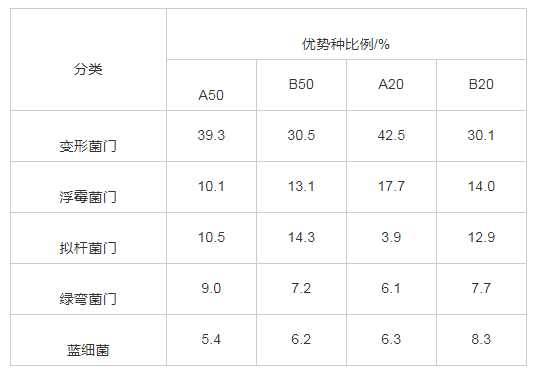
根据表2,在A、B区,不同采样深度也存在明显差异,A20样品中变形菌门、浮霉菌门丰度分别是42.5%和17.7%,均高于A50的丰度值。每个点的样品微生物群落组成在其他门水平上也存在较大差异。拟杆菌门A区的相对丰度均低于B区,同时B50采样点的丰度大于B20。
表2 不同位点纤维基质上优势物种的分布比例
3、 小结
Alpha多样性反映的是单个样品的物种多样性,包括observed species、Chao1,Shannon及Simpson指数等。样品不同的Alpha多样性指数的数值中,observed species和Chao1指数反映了样品中群落的丰度,即单指群落中物种数量,而不考虑群落中每个物种的丰度情况[7]。Shannon及Simpson指数反映群落的多样性,受样品群落中物种丰富度和物种均匀度的影响。在物种丰富度相同的情况下,群落中各物种具有越大的均匀度,则认为群落具有越大的多样性。Shannon和Simpson指数值越大,说明个体分配越均匀。若每一个体都属于不同的种,Shannon和Simpson指数就大;若每一个体都属于同一种,则Shannon和Simpson指数就小[7]。
在纤维基质上,A50处的Chaol和observed species指数略高于B50,Shannon和Simpson指数也均呈相同趋势,说明种植植物区样品的群落的丰度和多样性高于无植物区。同时还发现,在两处试验点50 cm的Shannon和Simpson指数均大于20 cm处指数,说明在50 cm处物种多样性相对较高。
物种丰度的分析是对每个OTU进行物种分类。结果表明,变形菌门、浮霉菌门在每个样品中均占有绝对优势,且这2种优势种总的比例在种植植物区整体略高于无植物区,而拟杆菌门和绿弯菌门在植物区均是A50>A20,在无植物区没有明显差异。
参考文献
[1] 宋庆辉,杨志峰.对我国城市河流综合管理的思考[J].水科学进展,2002,13(3):377-382.
[2] 王洁,王忠凯,李章林,等.现浇绿化混凝土护坡在农村河道整治工程中的应用[J].水利技术监督,2019(2):221-223.
[3] 高怡宁,陈锴新,余诗怡,等.温州市境内城市河流底泥氨氧化菌富集培养物微生物群落结构分析[J].生物学通报,2018,45(8):1676-1684.
[4] 袁亦舟,张伟国,徐建中.青稞酒曲微生物多样性分析及米根霉制曲条件优化[J].食品与发酵工业,2018,44(5):39-45.
[5] MASELLA A P,BARTRAM A K,TRUSZKOWSKI J M,et al.PANDAseq:paired-end assembler for illumina sequences[J].BMC Bioinformatics,2012,13(1):31.
[6] EDGAR R C.UPARSE:highly accurate OTU sequences from microbial amplicon reads[J].Nature Methods,2013,10(10):996.
[7] PAUL F K,JOSEPHINE Y A.Bacterial diversity in aquatic and other environments:what 16S rDNA libraries can tell us[J].FEMS Microbiol Ecol,2004,47:161-177.





