
早在 1960 年,Lave 和 Green[1]就发现了小鼠中 Atrn 突变基因能够抑制灰鼠色基因座( agouti)的表达,阻止黄色毛的形成。随着分子生物技术的不断完善及各个相关领域研究的不断深入,近几年来,通过对 Atrn 进行遗传分析发现: Atrn 为常染色体隐性基因,抑制小鼠黄色毛发的形成[2]; 最近也发现 Atrn 和 Mgrn 基因丧失功能突变小鼠能引起进行性的神经组织海绵状变性[3 -4],同时显示 Atrn 能够抑制 Agouti 而不是AGRP 所引起的肥胖; 分子结构与支持细胞或轴突导向密切相关,可能在中枢神经的形成和( 或)功能中起关键作用[5],Atrn 变异的小鼠的神经变性也伴有行为的异常: 年幼的小鼠有时出现轻微的震颤,较老的小鼠活动过度,有的则会发生神经肌肉衰弱和共济失调[6 -7]; Atrn 基因产生一个具有许多细胞外受体特征的跨膜蛋白[2,8 -9].由此看来,Atrn 基因在生物界的作用非常广泛,值得更进一步的探讨与研究。
应用生物信息学方法对 Atrn 基因的基因编码区进行预测和分析,研究 Atrn 基因在不同物种间和种内的遗传分化,为今后动物毛色、动物肥胖、动物神经中枢系统等问题以及动物遗传育种研究提供基础资料。
1 材料与方法
1. 1 序列来源
从 GenBank 中下载[10]人、牛、野牦牛、小须鲸、水牛、狼、狨、狨鼠、白犀牛、星鼻鼹鼠、犰狳、小马岛猬、埃氏象鼩、猫、裸隐鼠、食蟹猴、猕猴、雪貂、小家鼠、长臂猿、中央狐蝠、褐家鼠、野猪、松鼠 24 个物种 61 条 Atrn 基因序列( 表 1) .
1. 2 方法
将序列下载后,利用 BioEdit 软件对已下载的 24 个物种的 61 条 Atrn 基因共有片段长度为2 620 bp 的 CDS 序列进行多重比对分析,对比结果用 DanSP5 软件进行遗传多态性分析,生成单倍型,计算出物种间的核苷酸歧异度、平均核苷酸差异数、遗传分化指数。应用 MEGA6 软件的Neighbor-joining 方法构建系统进化树[11].利用蛋白分析专家 EXPASY 工具里的 Sig-nalP 4. 0 Server 对 Atrn 氨基酸序列进行预测,利用在线工具 TMHMM 2. 0 Server 对氨基酸序列的跨膜结构域进行预测,利用在线工具 ProtScale 对氨基酸序列的疏水性/亲水性进行预测,用 PBILLYON-GERLAND 信息库对氨基酸序列的二级结构进行预测,用 ProtParam 在线工具分析基因编码的氨基酸序列[12 -13].
2 结果与分析
2. 1 不同物种 Atrn 基因核苷酸多样性
2. 1. 1 多态位点、单倍型及核苷酸多样性通过对 24 个物种的 61 条序列比对结果进行分析,共发现 899 个多态位点,占 34. 31% ,其中单一多态位点 77 个,简约多态位点 822 个; 共31 种单倍型,单倍型的多样性为 0. 973,可以说明 Atrn 基因种间和种内变异较大。排除单一序列物种,对两个序列以上的 17 个物种进行分析( 表 2) ,人 Atrn 基因平均核苷酸差异数和核苷酸多样性最高,与其他物种存在的差异较大,这可以表明人的 Atrn 基因存在丰富的遗传多样性。【1】

2. 1. 2 不同物种 Atrn 基因的遗传分化根据表 3,各种群间的核苷酸歧异度和遗传分化的变化范围都很大,核苷酸歧异度( Dxy) 在0. 009 ~ 0. 133 之间,遗传分化 ( Gst) 在 0. 089 ~1. 000 之间,其中牛与水牛之间 Dxy 为 0. 009,Dxy 最小,说明它们的亲缘关系最近; 其次为人与长臂猿,Dxy 为 0. 016,人与食蟹猴的 Dxy 为0. 020,说明人类、长臂猿、食蟹猴的亲缘关系较近; 而犰狳与所有其他物种所分析的核苷酸歧异度相差很大,因此犰狳与其他物种的亲缘关系较远。根据物种间的核苷酸歧异度( Dxy) 构建的分子聚类图( 图 1) ,也可以得到类似的结果。
2. 1. 3 不同物种 Atrn 的( G + C) 含量一般来说,亲缘关系近的物种的核苷酸的碱基组成中具有相似的( G + C) 含量。经分析 17个物种 Atrn 的碱基组成,发现他们之间的( G + C)含量在 46. 10% ~52. 40%之间( 表 4) ,该结果再次说明 Atrn 在不同物种间发生一定程度的遗传变异,同时也说明了所研究物种的亲缘关系远近。
其中犰狳 Atrn 中( G + C) 含量为 52. 40%( 表 4) ,与其他物种 Atrn 中( G + C) 含量相差最大,说明犰狳与所研究的其他物种亲缘关系最远,与之前分析的结果相符。
2. 1. 4 同义替换和非同义替换不同物种 61 条 Atrn 基因序列编码区中同义替换平均位点数为 589. 40 个,非同义替换平均位点数为 2 029. 60 个。不同物种同义替换位点数( SS) 为 582. 33 ~621. 67( 表 4) ,同义替换核苷酸多样性均值[Pi( s) ]为 0. 148.非同义替换位点数( NSS) 为 1 997. 33 ~ 2 036. 67,非同义替换核苷酸多样性均值[Pi( a) ]为 0. 055( 表 4) .裸隐鼠 Atrn 基因编码区的非同义替换较其他物种高,其次是狼。如果某基因在种系之间,同义和非同义替换之比不同,则说明它在进化过程中所发生的突变并非中性,不符合中性突变理论[14].
若各同源基因间的同义和非同义替换率之比不同,其变化范围较大( 0. 011 6 ~0. 043 2) ,表明该基因在进化过程中不遵守中性理论且经历了净化选择[15].【2】
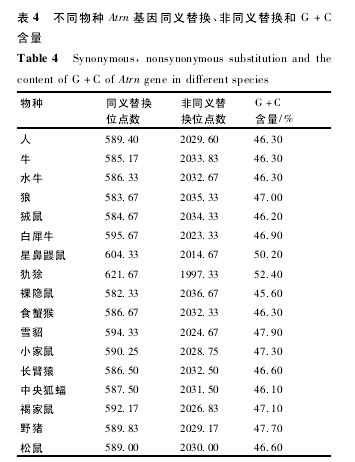
本研究发现,所选物种的 Atrn 基因的非同义替换位点数均明显高于同义替换位点数,说明所分析的这两个种群进化过程中可能经历了净化选择。
2. 2 不同物种氨基酸序列的预测与分析
2. 2. 1 信号肽及跨膜结构域信号肽位于分泌蛋白的 N 端,判断蛋白质是否含有信号肽部分,可以初步推测该蛋白质是否为分泌蛋白[17].利用在线工具 SignalP 4. 0 Serv-er 预测蛋白质的信号肽,发现 24 个物种中有 23个物种的 Atrn 蛋白质不含信号肽,但是猫的该蛋白质序列存在信号肽,可以初步判断猫 Atrn 编码的蛋白是分泌蛋白,其 C 值、S 值、Y 值在信号肽区域都较高( 图 2) .应用在线工具 TMHMM 2. 0Server 进行跨膜区预测,不同物种该蛋白均存在跨膜区,提示该蛋白是定位于生物膜的膜蛋白( 例如,图 3 中的矩形区域代表跨膜区) .总结以上结论可以推断 Atrn 蛋白质是属于定位在生物膜上的膜蛋白或分泌蛋白。
2. 2. 2 二级结构蛋白质二级结构预测是分子生物学中的重要问题。随着蛋白质序列数据的飞速增长,已知的蛋白质序列和已知的蛋白质结构之间的差距不断增大,蛋白质结构的预测变得越来越重要。
用 PBIL LYON-GERLAND 信息库对 Atrn 的氨基酸序列的二级结构进行预测,统计发现 24 个物种的基因编码的蛋白质序列主要是由无规卷曲( 58. 37% ~ 63. 75%) 和 α 螺 旋 ( 17. 53% ~23. 76% ) 构成,均不存在 β 折叠、β 转角。
2. 2. 3 氨基酸序列的组成成分及生化特性用 ProtParam 在线工具分析 Atrn 基因编码的氨基酸序列的组成成分及生化特性。Atrn 蛋白的不稳定系数在 43. 12 ~ 48. 40 之间,表明这种蛋白质不稳定[17].各氨基酸的理论 PI 为 7. 17,因此 Atrn 蛋白呈弱碱性,含量最多的是 Ser,占8. 2% ~ 9. 4% ; 其次是 Leu,占 6. 5% ~ 8. 0% ;第三是 Gly,占 6. 5% ~ 7. 8% .利用在线工具ProtScale 分析各个物种 Atrn 氨基酸序列,结果显示,本研究 24 个物种的 Atrn 氨基酸序列( 负数表现为亲水性,正值表现为疏水性) 最小值为- 3. 711,即亲水性最强; 最大值为 3. 800,即疏水性最强。总的来看,24 个物种的 Atrn 氨基酸序列亲水区域大于疏水区域,表现为亲水性,提示 Atrn 蛋白为亲水性蛋白,处于蛋白质分子的表面。
3 小结
不同物种间 Atrn 基因的核苷酸歧异度和单倍型间的遗传距离差异都较大,种内及种间遗传分化明显。Atrn 基因非同义替换位点数均明显高于同义替换位点数,Atrn 基因在进化过程中可能经历了净化选择。Atrn 基因物种间的亲缘关系与动物学分类相符。
Atrn 蛋白 N 端有信号肽和跨膜结构域,整个多肽链表现为亲水性,蛋白质二级结构的主要元件为无规则卷曲和 α-螺旋,Atrn 蛋白质属于定位在生物膜上的膜蛋白或分泌蛋白。
参考文献:
[1] Lane PW,Green MC. Mahogany,a recessive color mutationin linkage group V of the mouse[J]. Journal of Heredity ,1960,51: 228 - 230.
[2] Nagle DL,McGrail SH,Vitale J,Woolf EA,et al. The ma-hogany protein is a receptor involved in suppression of obesity[J]. Nature,1999,398( 6723) : 148 -152.
[3] He L,Gunn TM,Bouley DM,et al. A biochemical functionfor attractin in agouti induced pigmentation and obesity[J].Nature Genetics,2001,27 ( 1) : 40 - 47
[4] He L,Lu XY,Jolly AF,et al. Spongiform degeneration inmahoganoid mutant mice[J]. Science ,2003,299 ( 5607 ) :710 - 712.
[5] Lu X,Gunn TM,Barsh GS,et al. Distribution of Mahogany/Attractin mRNA in the rat central nervous system[J]. FEBSLetters,1999,462( 1) : 101 - 107.
[6] Dinulescu DM,Fan W,Boston BA,et al. Mahogany( mg)stimulates feeding and increases basal metabolic rate independ-ent of its suppression of agouti[J]. Proceedings of the NationalAcademy of Sciences of the United States of America,1998,95( 21) : 12707 -12712.
[7] Rehm S,Mehraein P,Anzil AP,et al. A new rat mutant withdefective overhairs and spongy degeneration of the central nerv-ous system: Clinical and pathologic studies[J]. Laboratoryanimal science ,1982,32 ( 1) : 70 - 73.
[8] Gunn TM,Miller KA,He L,et al. The mouse mahogany lo-cus encodes a transmembrane form of human attractin[J]. Na-ture,1999,398 ( 6723) : 152 - 156.
[9] Gunn TM,Barsh GS. Mahogany/attractin: Enroute from phe-notype to function[J]. Trends in Cardiovascular Medicine,2000,10( 2) : 7 - 81.
[10] 付鑫,李祥龙,周荣艳,等。 11 个物种 RPSA 基因编码区生物信息学分析[J]. 湖北农业科学,2012,51( 2) : 389- 390.
[11] 高红,彭静,沈一飞,等。 不同物种 FABP3 基因编码区及蛋白生物信息学分析[J]. 扬州大学学报: 农业与生命科学版,2014,35( 1) : 32.
[12] 杨洪一,孙立娜,张杰,等。 黄瓜绿斑驳花叶病毒抗原表位预测[J]. 河南农业科学,2013,42( 8) : 67 -70.
[13] 胡慧艳,贾青,陶隽,等。 家猪 TBP 蛋白结构与理化性质的生物信息学分析[J]. 河南农业科学,2014,43( 3) :128 - 132.
[14] Easteal S,Collet C. Consistent variation in amino-acid sub-stitution rate,despite uniformity of mutation rate: Protein e-volution in mammals is not neutral[J]. Molecular Biologyand Evolution,1994,11( 4) : 643 - 647.
[15] Mamirova L,Popadin K,Gelfand MS. Purifying selection inmitochondria,free-living and obligate intracellular proteobac-teria[J]. BMC Evolutionary Biology,2007,7 ( 17 ) : 17- 21.
[16] Liu DQ,Liu H,Shen HB. Predicting secretory signal se-quence cleavage sites by fusing the marks of global alignments[J]. Amino Acids,2007,32( 4) : 493 -496.
[17] Thorton JM. From genome to function[J]. Science,2001,292( 5524) : 2095 - 2097.





